Adventiv- und Rötelnviren in Priorix Tetra Batch A71CB256A
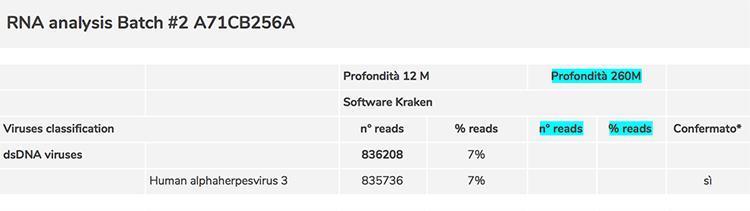
In der Screening-Phase für die Bewertung der Kontaminationen und Verunreinigungen des Priorix Tetra-Impfstoffs traten mehrere kritische Probleme auf: Das erste Problem betraf den Nichtnachweis des Rötelnvirus mit dem verwendeten Sequenzierungsgrad; Da dieses Ergebnis den Zweifel aufkommen ließ, dass es sich um einen Fehler für das verwendete Verfahren handeln könnte, wurde der Sequenzierungsgrad erheblich erhöht, um eine sehr hohe Tiefe zu erreichen (260 Millionen produzierte Sequenzen). Das Rötelnvirus wurde somit in 114 Kopien nachgewiesen, was 0.00004% der Gesamtsequenzen entspricht, und durch manuelles Ablesen der Sequenzen konnte jede Fehlerquelle der verwendeten Software beseitigt und das Vorhandensein von Röteln in der Probe definitiv bestätigt werden.
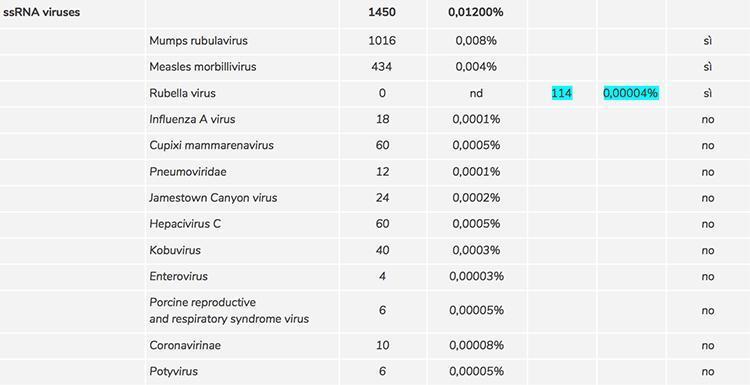
Dieses Verfahren ermöglichte es jedoch auch, die zufälligen Viren zu identifizieren, die in einer geringen Anzahl von Kopien vorhanden sind, und was wir gesehen haben, ist, dass die Anzahl der Kopien der zufälligen Viren die Anzahl der Kopien von Röteln überstieg. So entstanden zwei weitere sehr wichtige ungelöste Probleme:
- Ist die im Impfstoff vorhandene Rötel ausreichend, um eine immunogene Wirkung hervorzurufen, oder kann sie als Unterschwelle angesehen werden (dh als zufällige Kontamination)?
- Sind zufällige Viren wirklich vorhanden? Wenn ja, können sie gefährlich sein?
Zu Punkt 1) Wir können die Fähigkeit des abgeschwächten Rötelnvirus, als immunogenes Antigen zu wirken, aufgrund der vernachlässigbaren Anzahl von Kopien und der Abschwächung, die seine Wirksamkeit weiter schwächt, stark in Frage stellen, so dass wir es in jeder Hinsicht als Subthreshold-Kontaminante betrachten können
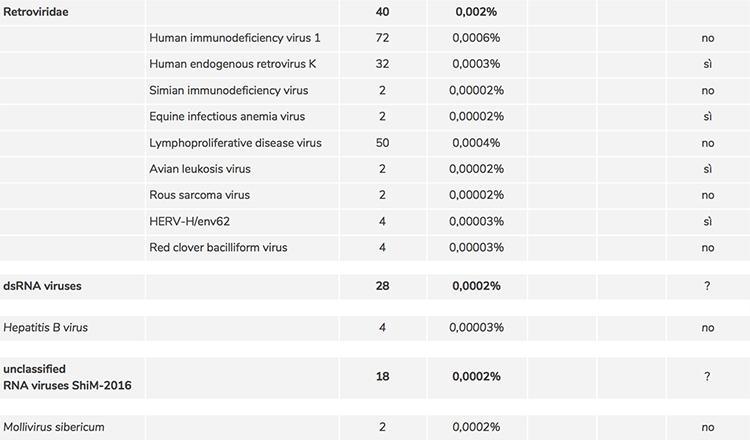
Zu Punkt 2) Die Liste der durch Screening entdeckten zufälligen Viren ergab nach Vergleich mit den in den offiziellen Datenbanken hinterlegten Sequenzen eine große Anzahl von Viren, deren Identifizierung aufgrund der mit der sehr geringen Anzahl verbundenen Unsicherheit von der Software, die sie klassifizierte, fälschlicherweise zugeordnet werden konnte von Kopien. Eine manuelle Überprüfung nacheinander war erforderlich, um ihr Vorhandensein mit einer anderen Software (BLAST) zu bestätigen, und somit konnte das Vorhandensein der folgenden kontaminierenden Retroviren bestätigt werden:
|
Humanes endogenes Retrovirus K |
32 Sequenzen |
|
Infektiöses Anämievirus für Pferde |
2 Sequenzen |
|
Vogel-Leukose-Virus |
2 Sequenzen |
|
HERV-H / env62 |
4 Sequenzen |
Es ist bekannt, dass diese Viren zufällige Kontaminanten des Impfstoffs sind, und ihre potenzielle Gefahr ist ebenfalls bekannt, weshalb die Hersteller ihre vollständige Abwesenheit im Impfstoff überprüfen müssen.
Daraus folgt, dass aus dieser eingehenden Analyse für diesen Impfstoff zwei Abweichungen in Bezug auf Wirksamkeit und Sicherheit hervorgehen:
- Das Vorhandensein in einer sehr geringen Anzahl von Kopien (Unterschwelle) von Röteln
- Das Vorhandensein potenziell gefährlicher zufälliger Viren
Was die Bestätigung von Spuren anderer Genome betrifft, die in den RNA-seq-Daten gefunden wurden, wie "nicht klassifizierte Bakterien- und Umweltviren" einschließlich Phagensequenzen, "dsRNA-Virus" und "nicht klassifizierte RNA-Viren ShiM-2016", die wenig bekannte virale Genome umfassen, u Eine zusätzliche eingehende Analyse ist erforderlich, um die richtige Zuordnung und effektive Präsenz nachzuweisen. Das Vorhandensein des Hepatitis B-Virus wurde durch die BLAST-Analyse NICHT bestätigt.
Nachstehend finden Sie eine Tabelle, in der die im Text beschriebenen Elemente zusammengefasst sind:
* Von der BLAST-Software manuell bestätigt
Herunterladen: CORVELVA-In-Depth-Analysis-of-Rubella-Virus-und-Virus-Sequenzen-Priorix-Tetra.pdf
Übersetzt vom CLiVa-Team - www.clivatoscana.com

