Vaccinegate: Séquençage complet du génome de MRC-5 contenu dans Priorix Tetra

Ces dernières analyses ont été possibles grâce à la contribution active des associations françaises Association Liberté Informations Santé (ALIS), Ligue nationale pour la liberté des vaccins (LNPLV) et l'association australienne Réseau australien des risques de vaccination (AVN) que nous remercions.
Les séquenceurs de nouvelle génération sont devenus des instruments de choix pour des analyses approfondies dans le domaine de la biologie et de la médecine, notamment la précision. Ces outils permettent une approche nouvelle et plus globale d'une série d'applications telles que le séquençage de novo, les études de métagénomique, l'épigénomique, le séquençage du transcriptome et le reséquençage du génome.
Cette dernière application (re-séquençage) est très répandue dans le domaine humain tant à des fins de recherche que de diagnostic et consiste en le séquençage avec la technologie NGS (Next Generation Sequencing) d'un génome individuel entier afin de cartographier des mutations nucléotidiques uniques (SNP, prononciation «snip»), des insertions et des suppressions de séquences plus ou moins longues se sont produites dans certaines positions du génome et des variations du nombre de copies de portions de génome / gènes (CNV, Copy Number Variants).
Cette procédure est utile pour comprendre les mécanismes de développement de certaines pathologies afin d'identifier les directions d'un futur traitement clinique, comme dans le cas du cancer. Avec cette méthode, en effet, l'héritage génétique d'un patient cancéreux peut être complètement décodé dans les tissus normaux et tumoraux, permettant de comprendre ce qui a changé dans le génome et, si possible, d'intervenir avec des protocoles ciblés.
La procédure de re-séquençage nécessite que l'ADN d'un individu soit mécaniquement cassé en petits fragments (400-500 paires de bases) et que les fragments soient liés à des voies d'ADN artificiel appelées adaptateurs, qui permettent de lier les fragments de ADN humain sur une surface en verre sur laquelle les bases sont ensuite lues (A, C, G, T). Les bases d'ADN sont lues par des réactions chimiques d'incorporation de nucléotides marqués avec des molécules fluorescentes. Les millions de séquences (lectures) obtenues par séquençage sur la surface du verre, sont ensuite cartographiées sur le génome humain de référence avec un logiciel approprié puis toutes les variantes présentes dans le génome analysé sont identifiées, par rapport à la référence.
Cette même procédure a été réalisée sur le génome humain présent dans le lot Priorix Tetra. n. A71CB256A, génome appartenant à la lignée cellulaire MRC-5 (d'origine fœtale); le travail a été effectué dans une entreprise située aux États-Unis, qui s'occupe systématiquement de l'analyse des génomes humains par reséquençage. *
* Le nom du laboratoire qui a effectué cette analyse sera repris dans la prochaine déclaration que nous déposerons auprès du parquet de Rome ainsi que des organes de contrôle italiens et européens. Les réalités qui déposent les résultats des analyses financées par Corvelva seront également immédiatement mises à jour avec ces résultats déroutants. Nous ne nions pas que, en tant que parents en premier lieu, nous sommes affligés par les résultats que nous rapportons ci-dessous - si ce que nous avons découvert jusqu'à présent n'était pas suffisant.
Résultats
Le génome humain de référence s'est avéré être couvert par des lectures provenant de l'ADN du vaccin pour 99.76%, donc pour la quasi-totalité de son intégralité. L'ADN humain fœtal représenté dans ce vaccin est donc un génome individuel complet ou est présent dans le vaccin à ADN génomique de tous les chromosomes d'un individu masculin (et en fait le fœtus dont dérive la lignée cellulaire MRC-5 est un homme).
Ci-dessous les résultats de l'analyse des différents types de variantes par rapport au génome humain de référence.
Variants nucléotidiques simples (SNP) et insertions / suppressions courtes (InDels)
Les variantes des bases d'ADN individuelles (SNP, prononcé «snip») sont des polymorphismes, c'est-à-dire des variations du matériel génétique, portées par un seul nucléotide. Les «InDels», d'autre part, sont de petites insertions et suppressions de moins de 50 pb de longueur et constituent une autre classe de variantes génomiques dans le génome humain.
Un total d'environ 3.6 millions de SNP ont été identifiés dans le génome du vaccin humain (dont 98.31% ont déjà été signalés dans la base de données publique dbSNP et 61.805 804 nouveaux ou originaux de cet ADN) et environ 89.42 85.106 InDels (dont XNUMX% déjà rapporté dans dbSNP et XNUMX nouveau).
La quantité de SNP est conforme à ce qui est rapporté dans la littérature dans un "génome humain typique" alors que les indels sont en quantité plus élevée que ce qui est rapporté par le "The 1000 Genomes Project Consortium", soit 800 mille contre 600 mille.
CNV (Copy Number Variants) et SV (Structural Variants)
Les variantes du nombre de copies (CNV) sont des variantes génomiques en raison des variations du nombre de copies de fragments relativement gros (plus de 50 pb) entre les génomes individuels. Il existe deux types de CNV: type "gain" (gain de copies) et type "loss" (perte de copies). 218 CNV ont été détectés dans le génome du vaccin humain, dont 82 de type "gain" (couvrant une partie du génome égale à environ 6.9 millions de paires de bases) et 136 CNV "de perte" (qui couvrent une partie du génome environ 70 millions de bases).
Comme décrit par le 1000 Genomes Project Consortium dans «Une référence mondiale pour la variation génétique humaine (Nature, vol. 526, 10 oct. 2015)», un génome humain typique contient de 2.100 2.500 à XNUMX XNUMX grandes variantes, notamment:
- 1.000 grandes suppressions
- 160 variations du nombre d'exemplaires (CNV)
- 10 inversions
qui influencent globalement, compte tenu également des insertions, sur 20 millions de bases de séquences.
Comme observé pour les INDEL courts, même dans le cas d'insertions et de suppressions importantes, le génome du vaccin n'est donc pas conforme à un génome humain "normal", étant beaucoup plus "réarrangé" qu'un génome d'un individu ordinaire.
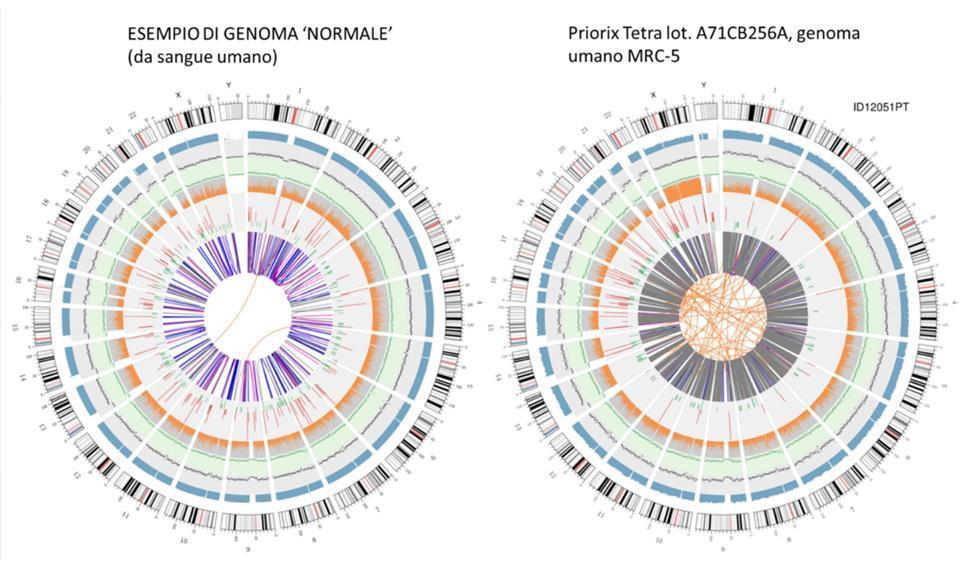
Visualisation circulaire du génome (intrigue circulaire)
Une représentation graphique du génome du vaccin appelé "intrigue circos" (qui est couramment utilisée pour représenter un génome reséquencé) est présentée ci-dessous, aux côtés d'une autre qui représente un génome re-séquencé à partir d'ADN extrait du sang d'un individu en bonne santé - génome "normal":
Signification des différents cercles concentriques
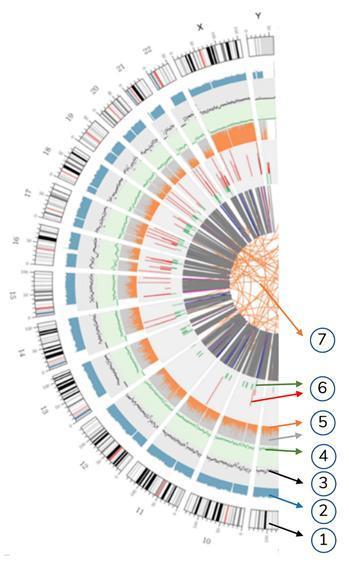
7) L'anneau le plus central représente l'inférence des SV (variantes structurelles) dans les régions exoniques et d'épissage. ENTRE (orange, translocations), INS (vert, insertions), DEL (suppressions, gris), DUP (duplications, rose) et INV (inversions, bleu).
6) Le sixième anneau représente l'inférence de CNV (Variantes en nombre d'exemplaires). Le rouge signifie gagner des morceaux d'ADN et le vert signifie perdre.
5) Le cinquième anneau représente la proportion de SNP en homozygotie (orange) et en hétérozygotie (gris) dans le style histogramme.
4) Le quatrième anneau (vert) représente la densité snp dans un style "graphique de dispersion".
3) Le troisième anneau (noir) représente la densité des INDEL dans un style "graphique de dispersion".
2) Le deuxième anneau (bleu) représente la couverture des lectures de style histogramme.
1) Le cercle extérieur (le premier cercle) est le nombre de chromosomes.
Une comparaison grossière peut également être faite entre l'ADN fœtal et l'ADN des cellules HeLa, la lignée cellulaire immortalisée également utilisée dans la production du vaccin contre la polio.
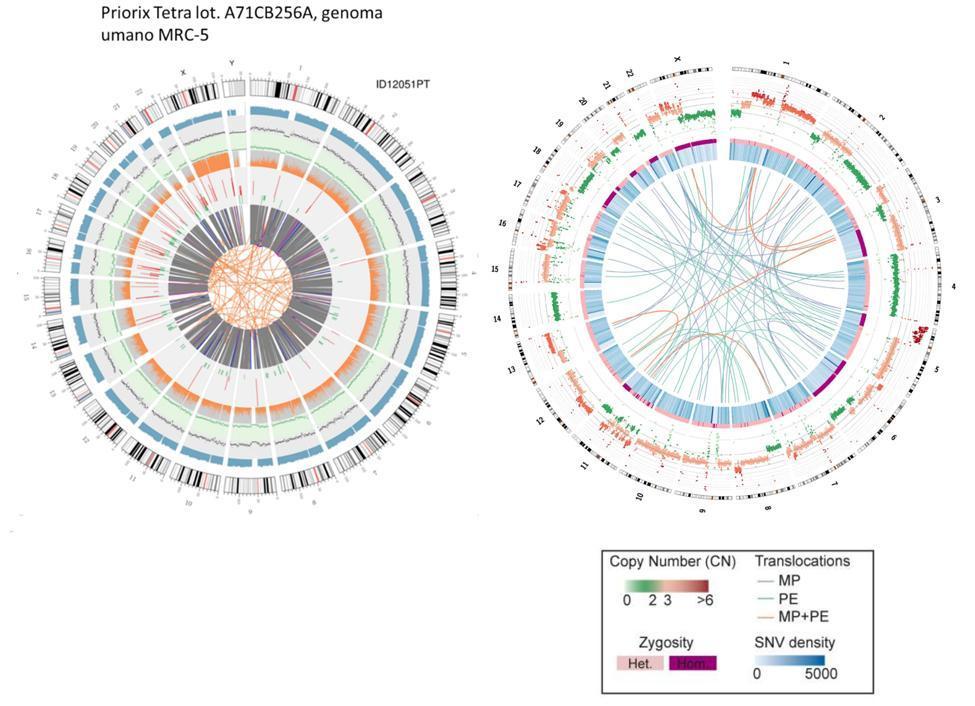
Il convient de souligner que les translocations des cellules HeLa représentées dans le tracé circos par les lignées de noyau se réfèrent à l'ensemble du génome (donc partie codante et non codante), alors que dans le cas des cellules vaccinales fœtales, elles ne se réfèrent qu'aux gènes codants.
Il n'est pas nécessaire d'être un scientifique pour comprendre du circos, simplement d'un coup d'œil, que le génome du vaccin n'est pas un génome qui peut être défini comme «normal». Les lignes oranges entrelacées au centre du circos, moins nombreuses dans l'anneau correspondant du génome «normal», nous font déjà comprendre l'anomalie de ce génome.
Conclusions
L'ADN génomique humain contenu dans le vaccin lot Prorix. n. A71CB256A est évidemment anormal, présentant des incohérences importantes par rapport à un génome humain typique, c'est-à-dire celui d'un individu sain. Il existe de nombreuses variantes inconnues (non notées dans les bases de données publiques) et plusieurs d'entre elles sont localisées dans des gènes impliqués dans le cancer. Ce qui est également manifestement anormal est l'excès de génome qui montre des changements dans le nombre de copies (CNV) et les variantes structurales (SV), telles que les translocations, les insertions, les suppressions, les duplications et les inversions, dont beaucoup impliquent des gènes.
La contribution potentielle des nombreuses variantes (non présentes dans la littérature scientifique et dans les bases de données publiques) au phénotype des cellules utilisées pour la croissance des virus vaccinaux n'est pas connue.
Dans le PDF, vous trouverez toutes les informations liées également aux implications pour la santé et la correspondance avec l'EMA.
Télécharger : CORVELVA-Ri-génome-séquençage-Priorix-humaine Tetra.pdf

