Priorix Tetra ludzki genom i linia komórkowa MRC-5 - badanie porównawcze

Krótka prezentacja wyników
Dzisiaj przedstawiamy kolejne i bardzo ważne dogłębne badanie genomowego sekwencjonowania linii komórkowej, które znaleźliśmy w czterowartościowej szczepionce MPRV (odra, różyczka, świnka i ospa wietrzna) Priorix Tetra® oraz linii komórkowej MRC-5 ( ten, który został użyty do opracowania samej szczepionki).
Podsumowując poprzednie opublikowane raporty:
- Po pierwsze, wykazaliśmy, że ilość DNA obecna w wyżej wymienionej szczepionce jest znacznie powyżej dopuszczalnego progu;
- Po drugie, zweryfikowaliśmy, że linia komórkowa jest faktycznie deklarowanym MRC-5; 2)
- Na koniec przeprowadziliśmy sekwencjonowanie genomu linii komórkowej bezpośrednio wyekstrahowanej ze szczepionki, podkreślając główne mutacje, które otwierają się na możliwość poważnego zagrożenia dla zdrowia przy stosowaniu tej szczepionki.
Prezentujemy tutaj dokładnie pracę porównawczą między dwoma genomami: tym, który faktycznie znaleźliśmy w szczepionce i tym, który został zakupiony w międzynarodowym banku linii komórkowych jako MRC-5.
W rezultacie obie linie wykazują istotne różnice z punktu widzenia stabilności genetycznej. W szczególności genom szczepionki jest znacznie zmodyfikowany w porównaniu z linią komórkową zarejestrowaną w 1966 roku.
Oznacza to, że producenci, w tym przypadku Priorix Tetra®, zakupili linię komórkową w optymalnych warunkach z punktu widzenia stabilności genetycznej i, z biegiem czasu ciągłego stosowania do celów produkcyjnych, wprowadzili na rynek szczepionki zawierające stopniowo coraz więcej zmodyfikowany ludzki materiał genetyczny, który jest niebezpieczny dla zdrowia samych osób zaszczepionych.
Ponieważ agencje regulacyjne nie wymagają okresowego monitorowania stabilności genetycznej linii płodowych, a nawet nie ustanawiają minimalnego progu bezpieczeństwa, możemy wywnioskować, że podstawowy wymóg dotyczący jakości i bezpieczeństwa szczepionek uzyskanych z ludzkich linii komórkowych nie został spełniony dla od jakiegoś czasu, a mianowicie konieczność sprawdzenia, czy nie rozwijają potencjalnie niebezpiecznych mutacji i rearanżacji oraz zapewnienia eliminacji przetwarzania pozostałości materiału genetycznego, jeśli są obecne.
Nie jesteśmy odpowiedzialni za określenie, która część procesu produkcji lub konserwacji mogłaby wpłynąć na jakość i bezpieczeństwo produktu, ale to, co możemy dziś potwierdzić, publikując ten raport, jest pilną koniecznością przeprowadzenia dogłębnej analizy wyników naszych analiz. powiadamiamy w ciągu ostatnich dwóch lat, koncentrując się na produkcie gotowym, sprzedawanym i podawanym, a nie tylko na pojedynczych zatwierdzonych „składnikach”.
Chcielibyśmy raz jeszcze przypomnieć, że produkty te są częścią włoskiego harmonogramu szczepień i są obecnie obowiązkowe wśród populacji pediatrycznej w naszym kraju, a także w innych krajach europejskich i ogólnie na całym świecie.
Organy regulacyjne ponoszą ogromną odpowiedzialność za bezpieczeństwo, które jest obecnie wyrażane słowami, ale w praktyce, o ile pokazują nasze analizy, nie można zagwarantować i w rzeczywistości wyraźnie nie jest ono zagwarantowane.
Cykliczne wyświetlanie genomów (wykres cyrkowy)
Graficzną reprezentację dwóch genomów zwanych „polem okrężnym” pokazano poniżej.
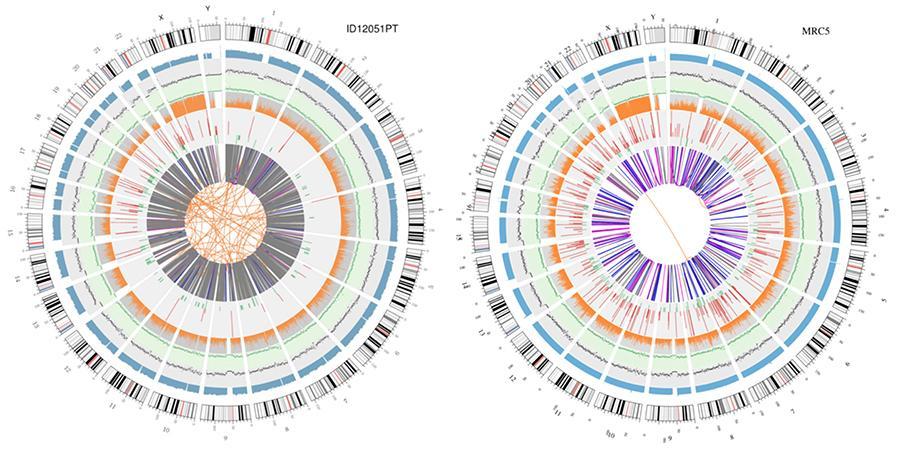
Wyjaśnienie różnych kół koncentrycznych
- Zewnętrzne koło (pierwsze koło) to liczba chromosomów.
- Drugi pierścień (niebieski) reprezentuje pokrycie odczytów w stylu histogramu. Każdy histogram to średnie pokrycie obszaru o wartości 0.5 Mb / s.
- Trzeci pierścień (czarny) przedstawia gęstość INDEL w stylu „wykresu dyspersji”. Każda czarna kropka jest obliczana jako liczba INDEL (małe wstawienia / usunięcia) w zakresie 1 Mbp.
- Czwarty pierścień (zielony) przedstawia gęstość snp w stylu „wykresu dyspersji”. Każda zielona kropka jest obliczana jako liczba SNP (wariantów pojedynczego nukleotydu) w zakresie 1 Mbp.
- Piąty pierścień przedstawia odsetek SNP w stylu histogramu homozygotyczności (pomarańczowy) i heterozygotyczności (szary). Każdy histogram jest obliczany na podstawie regionu 1 Mbp.
- Szósty pierścień reprezentuje wnioskowanie CNV (warianty liczby kopii). Czerwony oznacza zdobycie fragmentów DNA, a zielony oznacza utratę.
- Najbardziej centralny pierścień przedstawia wnioskowanie SV (Warianty Strukturalne) w obszarach heksagonalnych i splatanych. TRA (pomarańczowy, translokacje), INS (zielony, insercje), DEL (delecje, szary), DUP (duplikacje, różowy) i INV (inwersje, niebieski).
Wnioski
Te dwa genomy wykazują istotne różnice. W szczególności wydaje się, że genom szczepionki jest poważnie zmutowany w porównaniu z linią komórkową zdeponowaną w 1966 roku. Trzeba powiedzieć, że nie mieliśmy możliwości sekwencjonowania linii komórkowej używanej przez GlaxoSmithKline do produkcji szczepionki Priorix Tetra®, ale jeden z oryginalnych zapasów.
Firmy farmaceutyczne wykorzystujące linie komórkowe do produkcji leków i szczepionek mają własną linię MRC-5, ale fakt ten nie zmienia naszych wątpliwości, to znaczy, że nie ma okresowych wniosków o monitorowanie stabilności genetycznej i jest to poważny problem w naszym widok, szczególnie dlatego, że jest to dozwolone bez limitu progu.
Download: CORVELVA-Priorix-Tetra-ludzki-genom i-MRC-5-cell-line.pdf
Ludzki genom Priorix Tetra® i linia komórkowa MRC-5 - badanie porównawcze (standard ATCC MRC-5 ATCC® CCL-171 ™)
Obecność wirusa różyczki wykazano przez sekwencjonowanie biblioteki sekwencji RNA o wyjątkowo dużej głębokości (około 260 milionów wyprodukowanych sekwencji Illumina). Wykryto 114 sekwencji z 260 milionów, co stanowi 0.00004% wszystkich sekwencji. Sekwencje genomu różyczki zostały następnie potwierdzone ręcznie za pomocą oprogramowania BLAST (Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov/Blast.cgi). Biblioteka zsekwencjonowana na niższej głębokości (około 12 milionów zgodnych sekwencji Illumina, co równa się 6 milionom fragmentów zsekwencjonowanej biblioteki) nie wykryła ŻADNEGO odczytu, który można przypisać różyczce, w tej partii.
Inne sekwencje wirusa obecne w szczepionce (ospa wietrzna, odra i świnka) również zostały zatwierdzone w ten sam ręczny sposób, potwierdzając, że zostały poprawnie przypisane.
Wprowadzenie
Sekwencery nowej generacji stały się z wyboru narzędziami do szczegółowej analizy w dziedzinie biologii i medycyny, zwłaszcza precyzyjnej. Narzędzia te umożliwiają nowe i bardziej globalne podejście do szeregu zastosowań, takich jak sekwencjonowanie de novo, metagenomika, epigenomika, sekwencjonowanie transkryptomu i ponowne sekwencjonowanie genomu.
Ta ostatnia aplikacja (ponowne sekwencjonowanie) jest szeroko rozpowszechniona w dziedzinie ludzkiej zarówno do celów badawczych, jak i diagnostycznych i polega na sekwencjonowaniu za pomocą technologii NGS (Next Generation Sequencing) całego pojedynczego genomu w celu mapowania pojedynczych mutacji nukleotydowych (SNP, wymowa „ snip ”), insercje i delecje mniej lub bardziej długich sekwencji zachodziły w niektórych pozycjach genomu i zmiany liczby kopii części genomu / genów (CNV, warianty liczby kopii).
Procedura sekwencjonowania wymaga, aby DNA osobnika zostało mechanicznie rozbite na fragmenty o małych rozmiarach (400-500 pz) i te fragmenty są połączone z odcinkami sztucznego DNA zwanymi adapterami, które umożliwiają przymocowanie fragmentów ludzkiego DNA do szklanej powierzchni na których następnie odczytywane są zasady (A, C, G, T).
Odczyt DNA uzyskuje się poprzez włączenie reakcji chemicznych nukleotydów znakowanych cząsteczkami fluorescencyjnymi. Miliony sekwencji (odczytów), które są uzyskiwane przez sekwencjonowanie, wystąpiły na powierzchni szkła, są następnie mapowane na odniesienie do ludzkiego genomu za pomocą odpowiedniego oprogramowania, a następnie identyfikowane są wszystkie warianty obecne w analizowanym genomie w porównaniu do odniesienia.
Tę samą procedurę przeprowadzono na ludzkim genomie obecnym w partii Priorix Tetra®. A71CB256A i DNA wyekstrahowany z linii komórkowej MRC-5, zarejestrowany w 1966 r. W ATCC (MRC-5 ATCC® CCL-171 ™).
ATCC jest wiodącą na świecie organizacją zasobów i standardów materiałów biologicznych, której misja koncentruje się na pozyskiwaniu, uwierzytelnianiu, produkcji, konserwacji, rozwoju i dystrybucji mikroorganizmów odniesienia, linii komórkowych i innych materiałów o standardach biologicznych. Zostało założone w 1925 r., Kiedy komitet naukowców uznał potrzebę scentralizowanego zbioru materiałów biologicznych, który mógłby służyć naukowcom z całego świata.
Linia komórkowa MRC-5 pochodzi z normalnej tkanki płucnej od 14-tygodniowego płodu męskiego i została zdeponowana w ATCC przez JP Jacobs we wrześniu 1966 r. Zadeklarowano, że komórki te mogą się podwoić od 42 do 46 populacji przed starzeniem się. zaczyna się. Artykuł referencyjny dla tej linii komórkowej pochodzi z 1970 roku: https://www.nature.com/articles/227168a0
Efekt
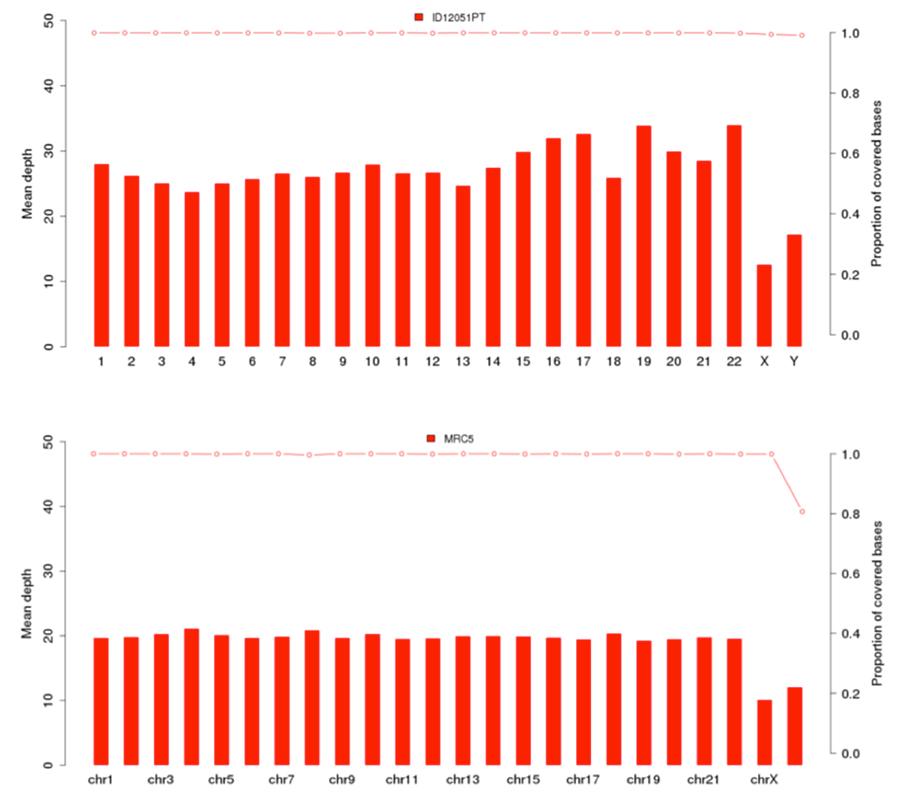
Jak wcześniej pokazano, ludzkie DNA obecne w partii Priorix Tetra®. n. A71CB256A (wskazany na poniższych wykresach jako „próbka ID12051PT”) jest kompletnym indywidualnym genomem, to znaczy, że genomowy DNA wszystkich chromosomów osobnika męskiego jest obecny. Wykresy pokrycia (średnia głębokość = średnie pokrycie) 22 ludzkich chromosomów oraz chromosomów X i Y pokazano poniżej, dla szczepionki (zdjęcie powyżej) i linii komórkowej MRC5 (zdjęcie poniżej).
Poniżej przedstawiono wyniki analizy różnych rodzajów wariantów.
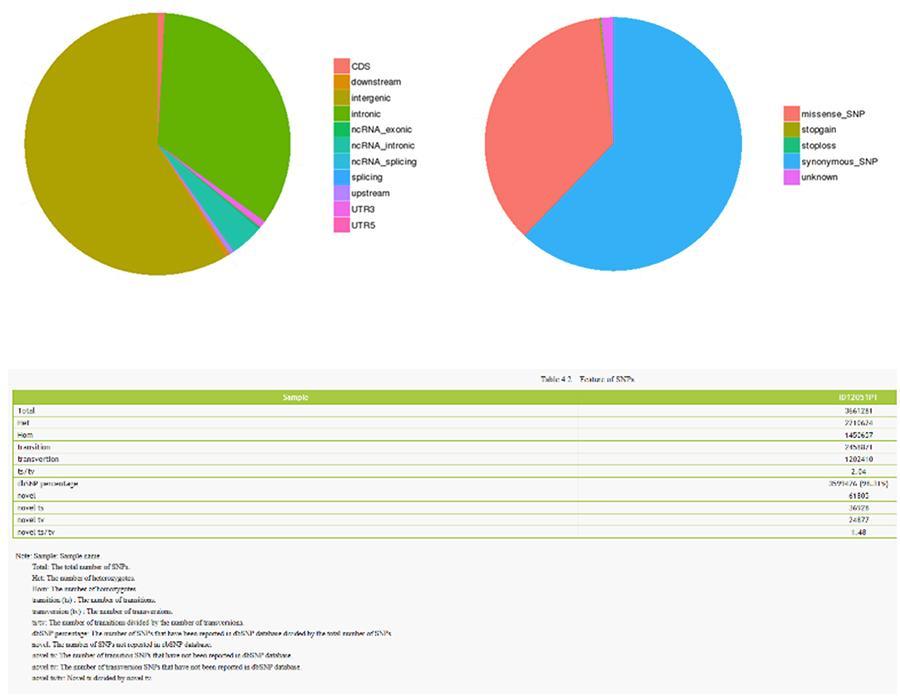
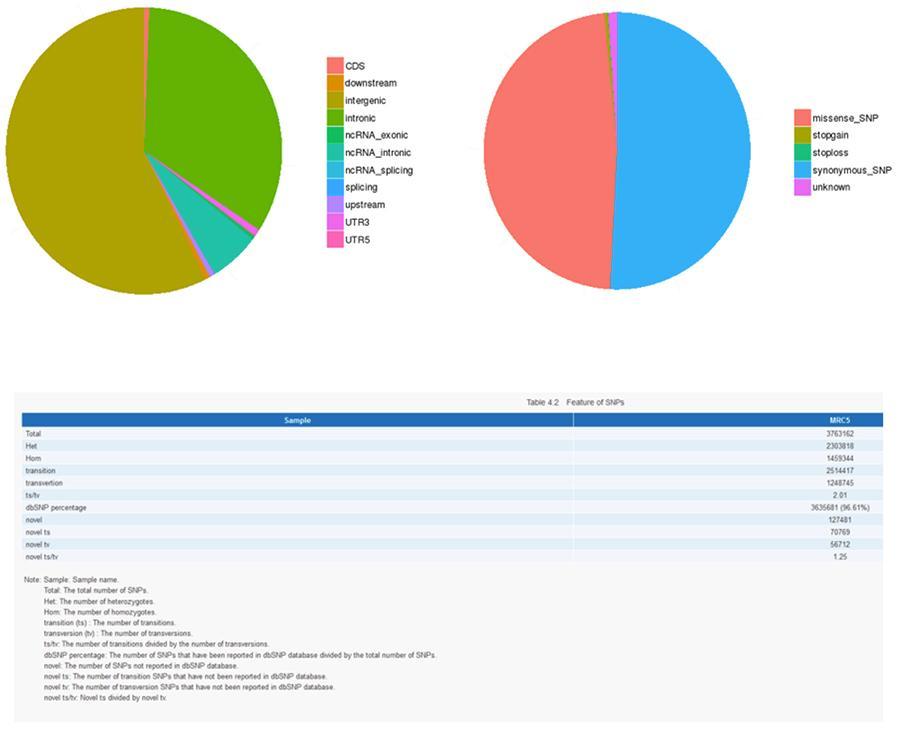
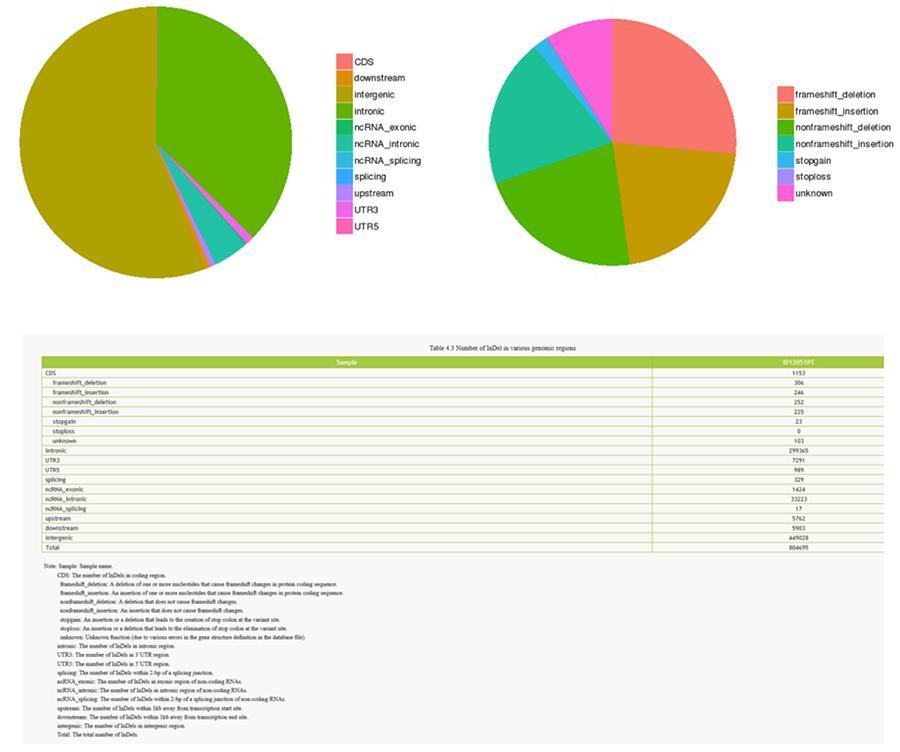
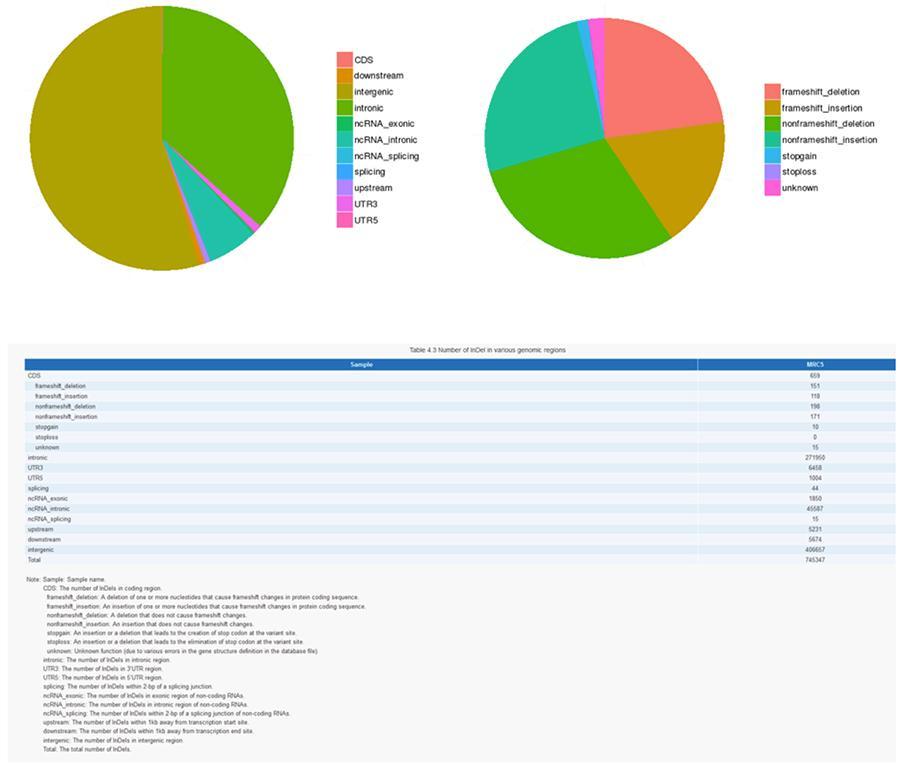
Polimorfizm pojedynczego nukleotydu (SNP) i krótkie insercje / delecje (InDels)
Warianty pojedynczego pojedynczego DNA (SNP, wymawiane „wycinanie”) są polimorfizmami, to znaczy odmianami materiału genetycznego przenoszonego przez pojedynczy nukleotyd. „InDels” to małe insercje i delecje o długości mniejszej niż 50 pz i stanowią kolejną klasę wariantów genomowych w ludzkim genomie.
SNP (polimorfizm pojedynczego nukleotydu) - ID12051PT
SNP (polimorfizm pojedynczego nukleotydu) - MRC-5
InDels (krótkie wstawianie / usuwanie) - ID12051PT
InDels (krótkie wstawianie / usuwanie) - MRC-5
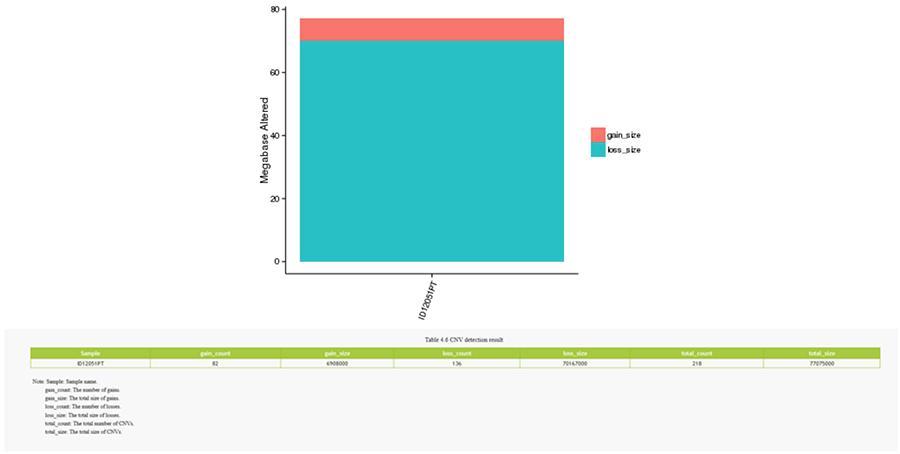
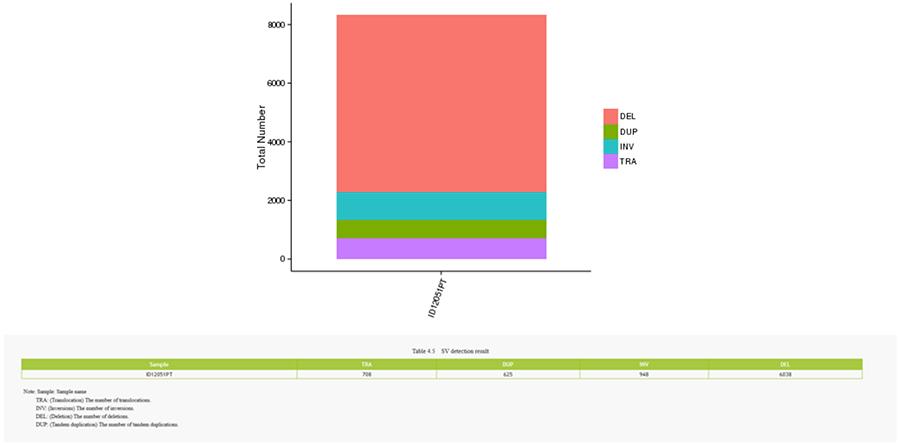
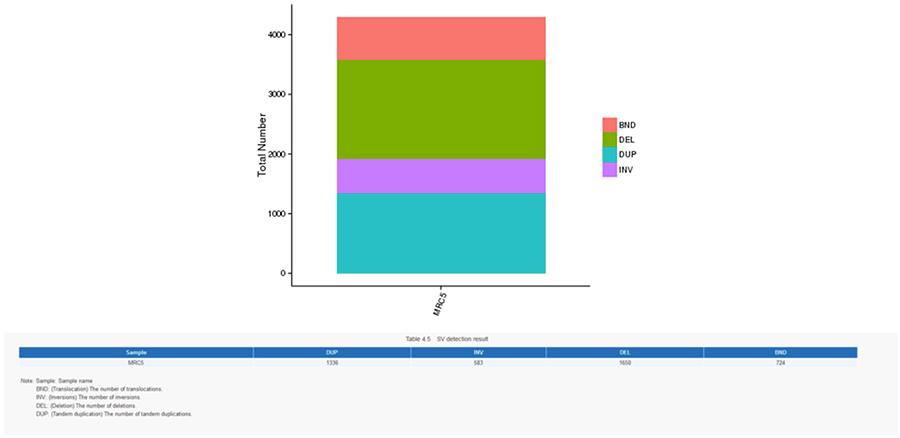
CNV (warianty numerów kopii) i SV (warianty strukturalne)
Warianty liczby kopii (CNV) są wariantami genomowymi z powodu zmian liczby kopii stosunkowo dużych fragmentów (dłuższych niż 50 pz) w poszczególnych genomach. Istnieją dwa rodzaje CNV: rodzaj „wzmocnienia” (wzrost liczby kopii) i rodzaj „utraty” (utrata kopii).
Warianty strukturalne (SV) to warianty genomowe o stosunkowo dużych wymiarach (> 50 pz), w tym delecje, duplikacje, insercje, inwersje i translokacje.
CNV (warianty numerów kopii)
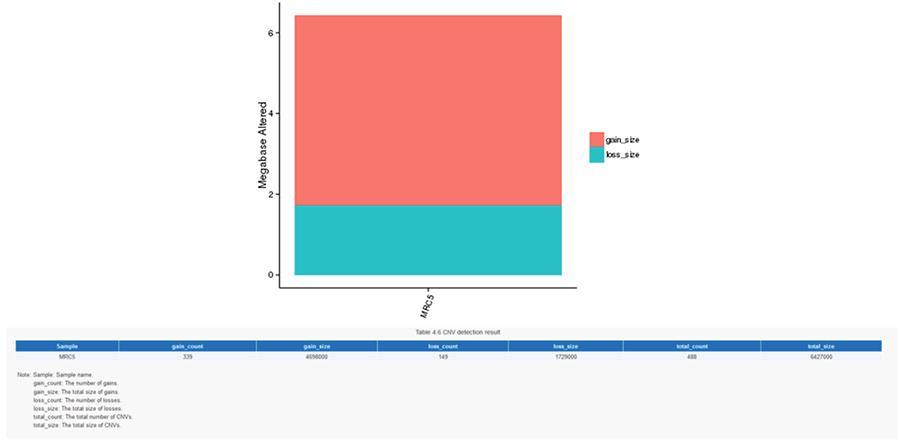
SV (warianty konstrukcyjne)
Kołowa wizualizacja genomów (wykres cyrkowy)
Graficzną reprezentację dwóch genomów (wykres cyrkowy) pokazano poniżej.
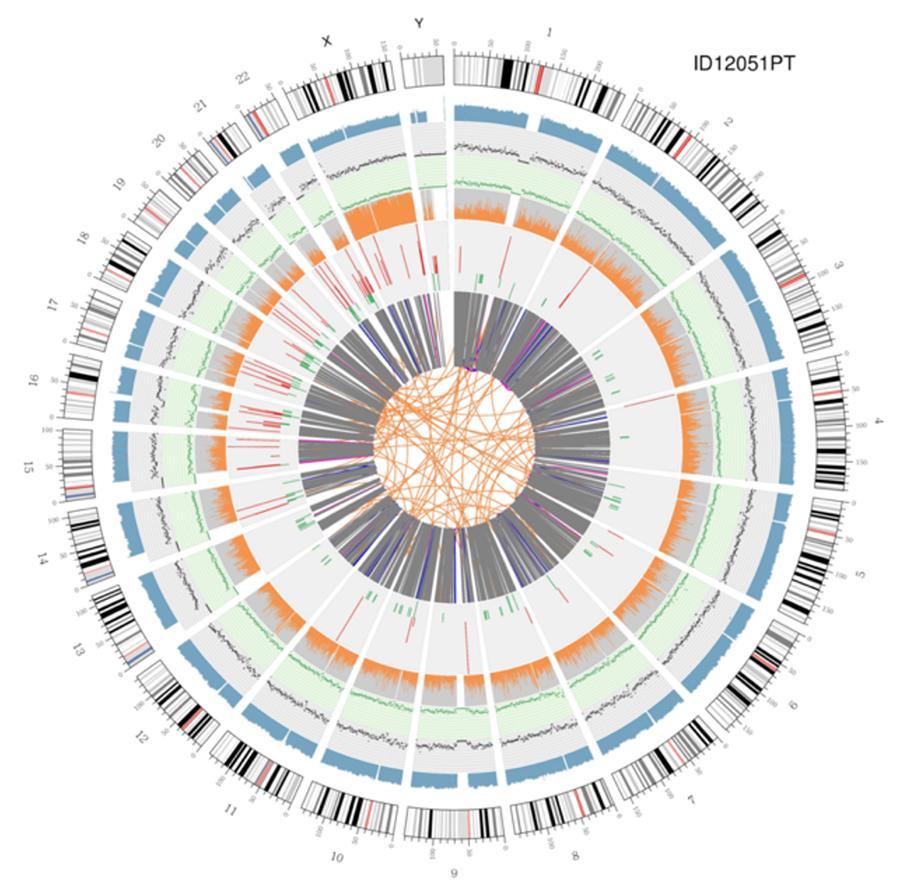
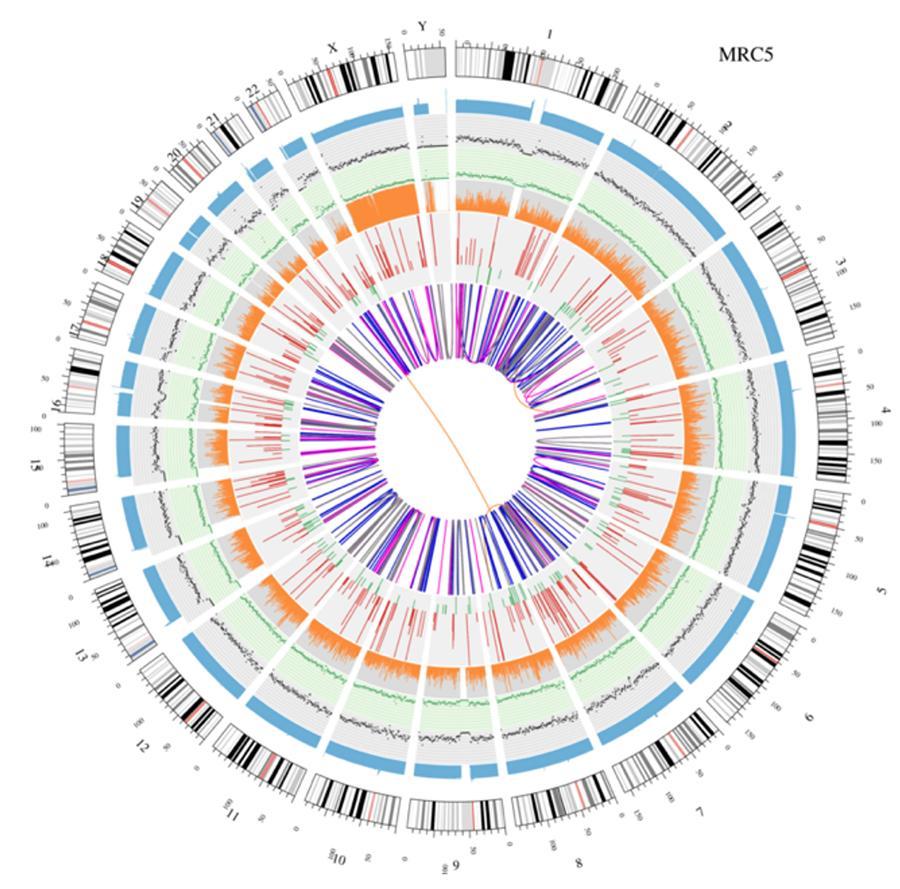
Znaczenie różnych kół koncentrycznych
- Zewnętrzny okrąg (pierwszy okrąg) to numer chromosomu.
- Drugi pierścień (niebieski) reprezentuje zasięg odczytów w stylu histogramu. Każdy histogram to średni zasięg w obszarze 0,5 Mbp.
- Trzeci pierścień (czarny) reprezentuje gęstość INDELI w „stylu grafu dyspersyjnego”. Każdy czarny punkt jest obliczany jako liczba PĘTLI (małe wstawienia / usunięcia) w zakresie 1 Mbp.
- Czwarty pierścień (zielony) reprezentuje gęstość snp w „stylu wykresu dyspersji”. Każda zielona kropka jest obliczana jako liczba SNP (polimorfizm pojedynczego nukleotydu) w zakresie 1 Mbp.
- Piąty pierścień reprezentuje proporcję homozygotycznego (pomarańczowego) i heterozygotycznego (szarego) SNP w stylu histogramu. Każdy histogram jest obliczany na podstawie regionu 1 Mbp.
- Szósty pierścień reprezentuje wnioskowanie CNV (warianty numerów kopii). Kolor czerwony oznacza wzrost kawałków DNA, a kolor zielony oznacza utratę.
- Najbardziej centralny pierścień reprezentuje wnioskowanie SV (warianty strukturalne) w obszarach egzonicznych i splicingowych. TRA (translokacja, pomarańczowy), INS (wstawki, zielony), DEL (usuwanie, szary), DUP (duplikacje, różowy) i INV (inwersje, niebieski).
wnioski
Te dwa genomy wykazują ważne różnice. W szczególności genom szczepionki okazał się mocno zmieniony w porównaniu z genomem linii komórkowej zdeponowanej w 1966 r. Należy powiedzieć, że nie mieliśmy możliwości przeprowadzenia sekwencjonowania linii komórkowej używanej przez GlaxoSmithKline do produkcji szczepionka Priorix Tetra®, ale oryginalna szczepionka. Firmy farmaceutyczne, które wykorzystują linie komórkowe do produkcji leków i szczepionek, mają własną linię MRC-5, ale to nie zmienia naszych podejrzeń, to znaczy, że fakt, że kontrola stabilności genetycznej nie jest okresowo wymagana, jest bardzo poważna, naszym zdaniem, szczególnie dlatego, że może występować bez progu granicznego.
wnioski
Dwa genomy wykazują ważne różnice. W szczególności genom szczepionki okazał się silnie zmieniony w porównaniu z linią komórkową zdeponowaną w 1966 r. Trzeba powiedzieć, że nie mieliśmy możliwości sekwencjonowania linii komórkowej wykorzystywanej przez GlaxoSmithKline do produkcji szczepionki Priorix Tetra, ale zapasów oryginalne. Firmy farmaceutyczne, które wykorzystują linie komórkowe do produkcji leków i szczepionek, mają swoją linię MRC-5, ale to nie zmienia naszych podejrzeń, to jest fakt, że kontrola stabilności genetycznej nie jest wymagana okresowo i jest to naszym zdaniem bardzo poważne , przede wszystkim dlatego, że może być obecny bez ograniczeń.

