Comparaison entre le génome humain contenu dans Priorix Tetra et dans la lignée cellulaire MRC-5

Brève présentation des résultats
Nous présentons aujourd'hui une autre étude très importante sur le séquençage génomique de la lignée cellulaire trouvée dans le vaccin quadrivalent MPRV (rougeole, rubéole, oreillons et varicelle) Priorix Tetra, et celle de la lignée cellulaire MRC-5 (qui est celle déclarée utilisée dans le développement de la vaccin lui-même).
Résumé des précédents rapports publiés:
- nous avons d'abord montré comment la quantité d'ADN présente dans le vaccin susmentionné était bien au-dessus du niveau autorisé;
- nous avons ensuite vérifié que la lignée cellulaire est en fait le MRC-5 déclaré;
- nous avons ensuite procédé à séquençage du génome de la lignée cellulaire extraite directement du vaccin, mettant en évidence des mutations très lourdes entraînant la possibilité de risques graves pour la santé lors de l'utilisation de ce vaccin.
Ce que nous présentons ici est précisément le travail de comparaison entre les deux génomes: ce que nous avons vraiment trouvé dans le vaccin et celui du MRC-5 acheté dans la base de données ATCC.
Le résultat est que les deux lignées présentent des différences importantes du point de vue de la stabilité génétique. En particulier, le génome du vaccin semble être fortement modifié par rapport à celui de la lignée cellulaire déposée en 1966.
Cela signifie que les producteurs, dans ce cas du Priorix tetra, ont acheté la lignée cellulaire dans des conditions optimales du point de vue de la stabilité génétique et avec une utilisation continue dans le temps pour la production ont mis sur le marché des vaccins contenant du matériel génétique humain progressivement de plus en plus modifié et dangereux pour la santé des vaccinés.
Étant donné que les organismes de réglementation n'exigent pas de contrôle périodique de la stabilité génétique des lignées fœtales et ne fixent même pas de seuil minimal de sécurité, nous pouvons en déduire qu'une exigence fondamentale pour la qualité et la sécurité des vaccins obtenues à partir de lignées cellulaires humaines, c'est-à-dire la nécessité de vérifier qu'elles n'acquièrent pas de mutations et de réarrangements potentiellement dangereux et de garantir l'élimination des résidus de traitement du matériel génétique le cas échéant.
Il ne nous appartient pas d'identifier quelle partie du processus de production ou de conservation peut affecter la qualité et la sécurité du produit, mais ce que nous pouvons témoigner aujourd'hui en publiant ce rapport, c'est qu'il est urgent que les organismes de contrôle procèdent à un examen approfondi des résultats que nos analyses ont mis en évidence. au cours de ces deux années, en se concentrant sur le produit fini, vendu et administré et pas seulement sur les "ingrédients" individuels autorisés.
Rappelez-vous encore une fois que ces produits font partie du calendrier de vaccination italien et sont actuellement administrés obligatoirement à la population pédiatrique de notre pays ainsi qu'à d'autres pays européens et en général partout dans le monde.
Les organes de contrôle ont une énorme responsabilité en matière de sécurité qui est actuellement annoncée par des mots mais qui, en fait, et comme il ressort de nos analyses, ne peut être garantie et est en effet clairement ignorée.
Visualisation circulaire des génomes (intrigue circulaire)
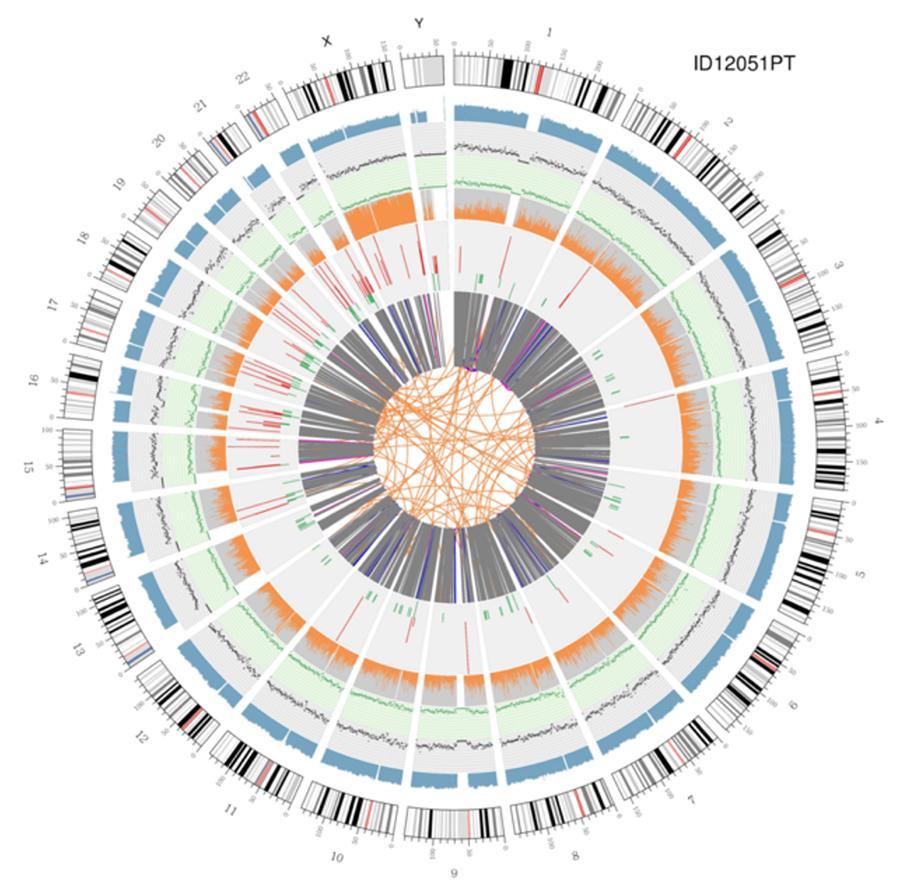
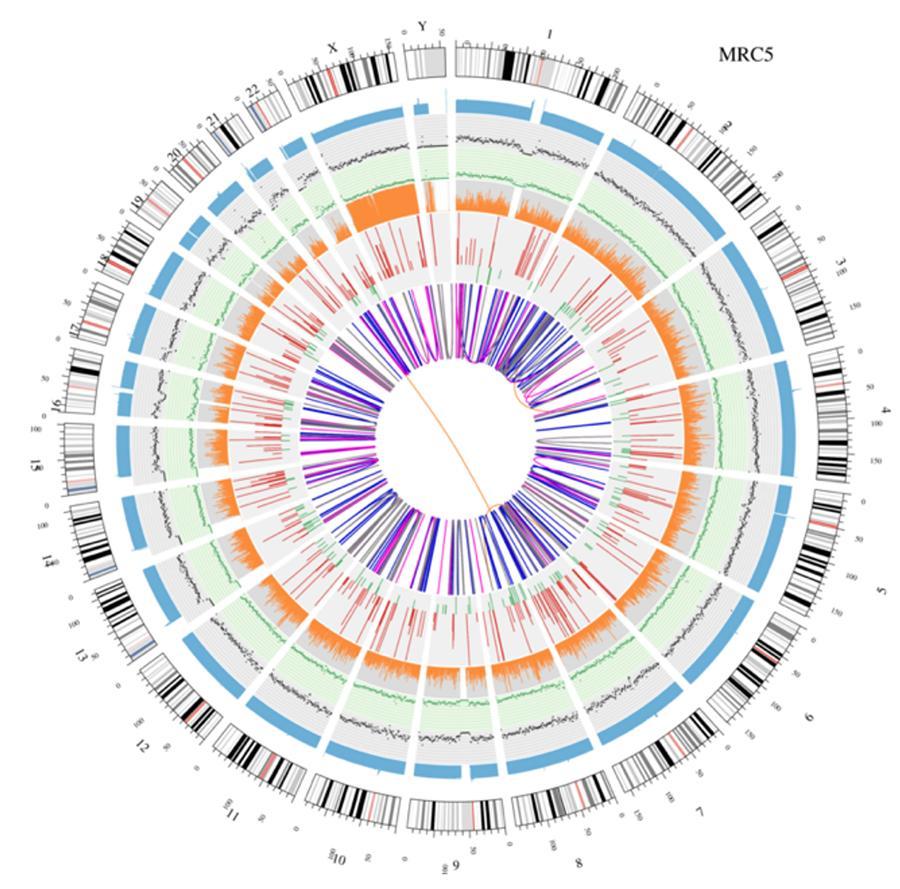
Une représentation graphique des deux génomes appelés «intrigue circos» est présentée ci-dessous.
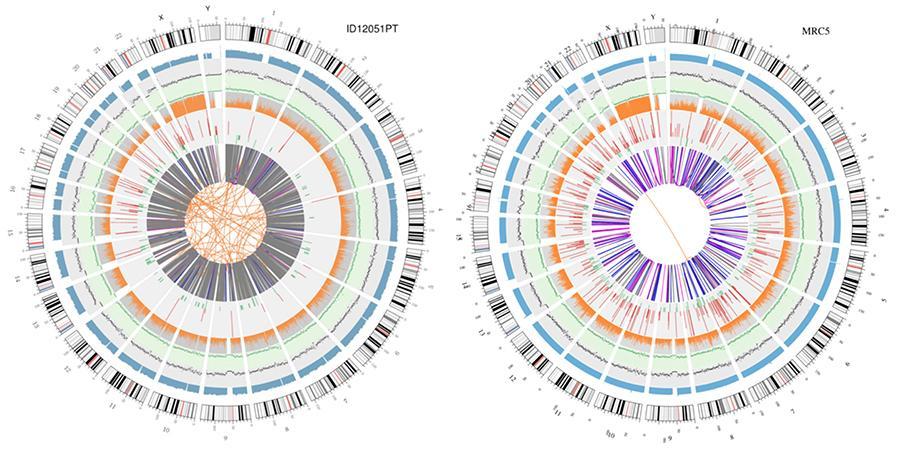
Signification des différents cercles concentriques
- Le cercle extérieur (le premier cercle) est le nombre de chromosomes.
- Le deuxième anneau (bleu) représente la couverture des lectures de style histogramme. Chaque histogramme est la couverture moyenne d'une zone de 0,5 Mbp.
- Le troisième anneau (noir) représente la densité des INDEL dans un style de «graphique de dispersion». Chaque point noir est calculé comme le nombre d'INDEL (petites insertions / suppressions) dans une plage de 1 Mbp.
- Le quatrième anneau (vert) représente la densité snp dans un style de «graphique de dispersion». Chaque point vert est calculé comme le nombre de SNP (variantes nucléotidiques simples) dans une plage de 1 Mbp.
- Le cinquième anneau représente la proportion d'homozygotie (orange) et d'hétérozygotie (gris) SNP dans le style histogramme. Chaque histogramme est calculé à partir d'une région de 1 Mbp.
- Le sixième anneau représente l'inférence de CNV (Variantes en nombre d'exemplaires). Le rouge signifie gagner des morceaux d'ADN et le vert signifie perdre.
- L'anneau le plus central représente l'inférence des SV (variantes structurelles) dans les régions exoniques et d'épissage. ENTRE (orange, translocations), INS (vert, insertions), DEL (suppressions, gris), DUP (duplications, rose) et INV (inversions, bleu).
Conclusions
Les deux génomes présentent des différences importantes. En particulier, le génome du vaccin se révèle fortement modifié par rapport à celui de la lignée cellulaire déposée en 1966. Il faut dire que nous n'avons pas eu la possibilité de séquencer la lignée cellulaire utilisée par GlaxoSmithKline pour la production du vaccin Priorix Tetra, mais celle du stock d'origine. Les sociétés pharmaceutiques qui utilisent des lignées cellulaires pour la production de médicaments et de vaccins ont leur lignée MRC-5 mais cela ne change pas nos soupçons, c'est le fait que le contrôle de la stabilité génétique n'est pas requis périodiquement et c'est très grave à notre avis , surtout parce qu'il est permis d'être présent sans limite.
Télécharger : CORVELVA-comparaison humaine génome-Priorix-Tetra-et-ligne-MRC-5.pdf
Comparaison entre le génome humain contenu dans Priorix Tetra et dans la lignée cellulaire MRC-5 (norme ATCC MRC-5 ATCC® CCL-171 ™)
La présence du virus de la rubéole a été démontrée par le séquençage d'une bibliothèque d'ARN-seq extrêmement profonde (environ 260 millions de séquences Illumina produites). 114 des 260 millions de séquences ont été détectées, soit 0.00004% de la séquence totale. Les séquences du génome de la rubéole ont ensuite été confirmées manuellement avec le logiciel BLAST (Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov/Blast.cgi). La bibliothèque séquencée à plus faible profondeur (environ 12 millions de séquences couplées Illumina, soit 6 millions de fragments de bibliothèque séquencés) n'avait détecté la présence d'aucune lecture attribuable à la rubéole, dans ce lot.
Les séquences des autres virus présents dans le vaccin (varicelle, rougeole et oreillons) ont également été validées de la même manière manuelle, confirmant qu'elles ont été correctement affectées.
Introduction
Les séquenceurs de nouvelle génération sont devenus des instruments de choix pour des analyses approfondies dans le domaine de la biologie et de la médecine, notamment la précision. Ces outils permettent une approche nouvelle et plus globale d'une série d'applications telles que le séquençage de novo, la métagénomique, l'épigénomique, le séquençage du transcriptome et le reséquençage du génome.
Cette dernière application (re-séquençage) est très répandue dans le domaine humain tant à des fins de recherche que de diagnostic et consiste en le séquençage avec la technologie NGS (Next Generation Sequencing) d'un génome individuel entier afin de cartographier des mutations nucléotidiques uniques (SNP, prononciation 'snip '), des insertions et des suppressions de séquences plus ou moins longues se sont produites dans certaines positions du génome et des variations dans le nombre de copies de portions de génome / gènes (CNV, Copy Number Variants).
La procédure de séquençage nécessite que l'ADN d'un individu soit mécaniquement brisé en petits fragments (400-500 paires de bases) et que des segments d'ADN artificiel appelés adaptateurs soient liés aux fragments, ce qui permet de lier les fragments d'ADN humain à une surface vitrée sur laquelle les bases sont ensuite lues (A, C, G, T). Les bases d'ADN sont lues en incorporant des réactions chimiques de nucléotides marqués avec des molécules fluorescentes. Les millions de séquences (lectures) obtenues par séquençage sur la surface du verre sont ensuite cartographiées sur le génome humain de référence avec un logiciel approprié puis toutes les variantes présentes dans le génome analysé par rapport à la référence sont identifiées.
Cette même procédure a été réalisée sur le génome humain présent dans le lot Priorix Tetra. n. A71CB256A et sur l'ADN extrait de la lignée cellulaire MRC-5, déposé en 1966 chez ATCC (MRC-5 ATCC® CCL-171 ™).
ATCC est la principale organisation mondiale de ressources et de normes en matière de matériaux biologiques dont la mission se concentre sur l'acquisition, l'authentification, la production, le stockage, le développement et la distribution de micro-organismes de référence, de lignées cellulaires et d'autres matériaux biologiques standard. Il a été créé en 1925, lorsqu'un comité de scientifiques a reconnu la nécessité d'une collection centralisée de matériel biologique pouvant servir les scientifiques du monde entier.
La lignée cellulaire MRC-5 est dérivée du tissu pulmonaire normal d'un fœtus mâle de 14 semaines et a été déposée dans l'ATCC par JP Jacobs en septembre 1966. Ces cellules seraient capables de doubler 42 à 46 populations avant l'initiation. de sénescence. L'article de référence pour cette lignée cellulaire remonte à 1970: https://www.nature.com/articles/227168a0
Résultats
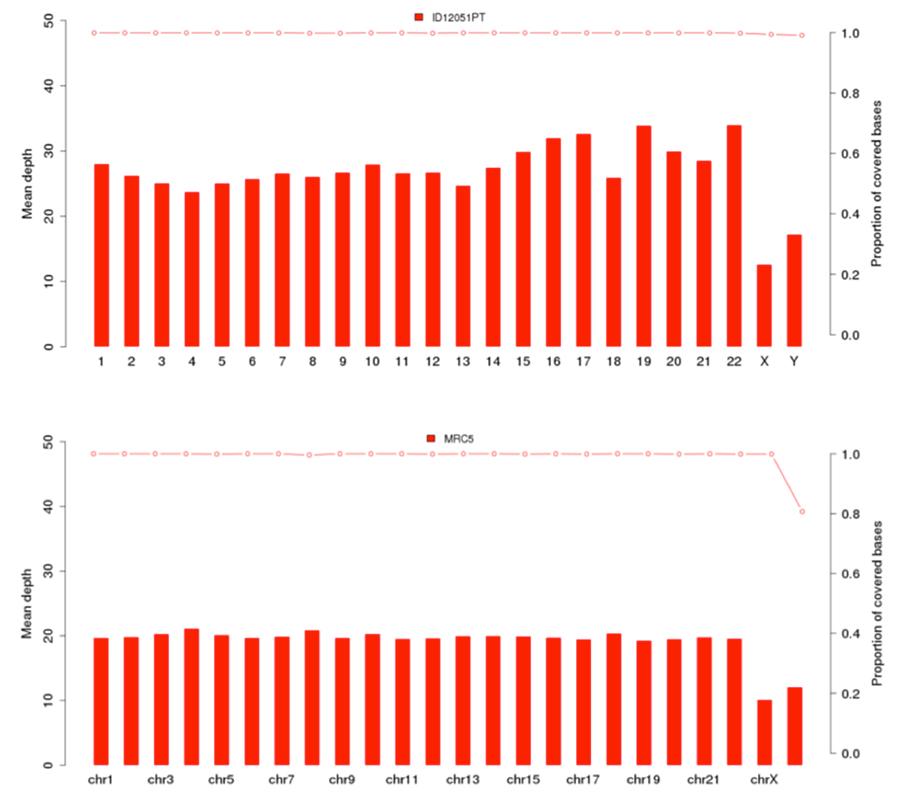
Comme indiqué précédemment, l'ADN humain présent dans le lot Priorix Tetra. n. A71CB256A (indiqué dans les graphiques suivants comme échantillon ID12051PT) est un génome individuel complet, c'est-à-dire qu'il existe de l'ADN génomique de tous les chromosomes d'un individu masculin. Les graphiques de couverture (profondeur moyenne = couverture moyenne) des 22 chromosomes humains et des chromosomes X et Y sont présentés ci-dessous, pour le vaccin (image ci-dessus) et la lignée cellulaire MRC5 (image ci-dessous).
Les résultats de l'analyse des différents types de variantes sont présentés ci-dessous.
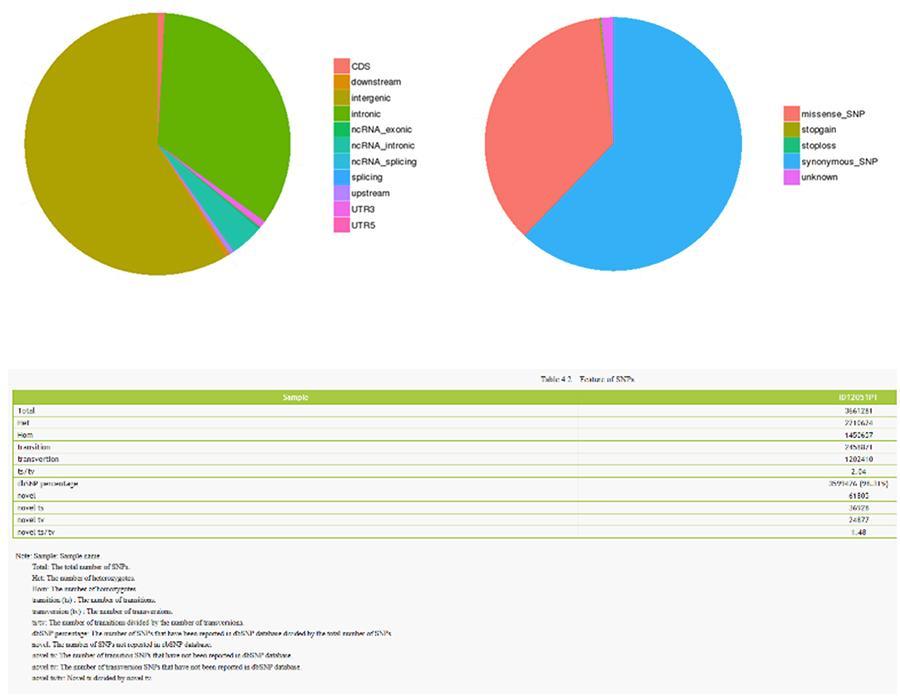
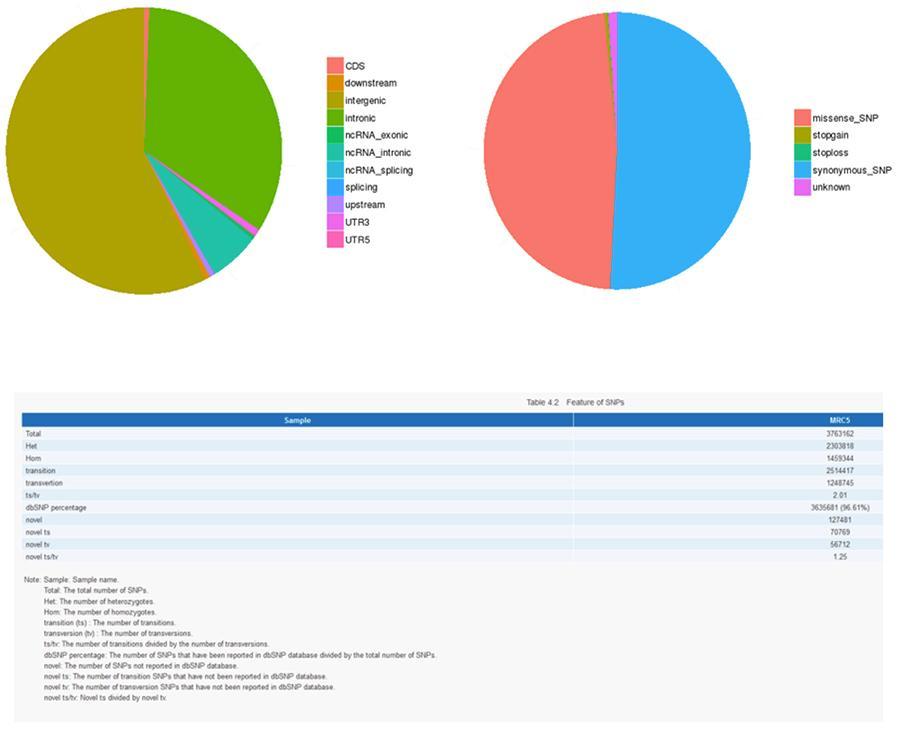
Variants nucléotidiques simples (SNP) et insertions / suppressions courtes (InDels)
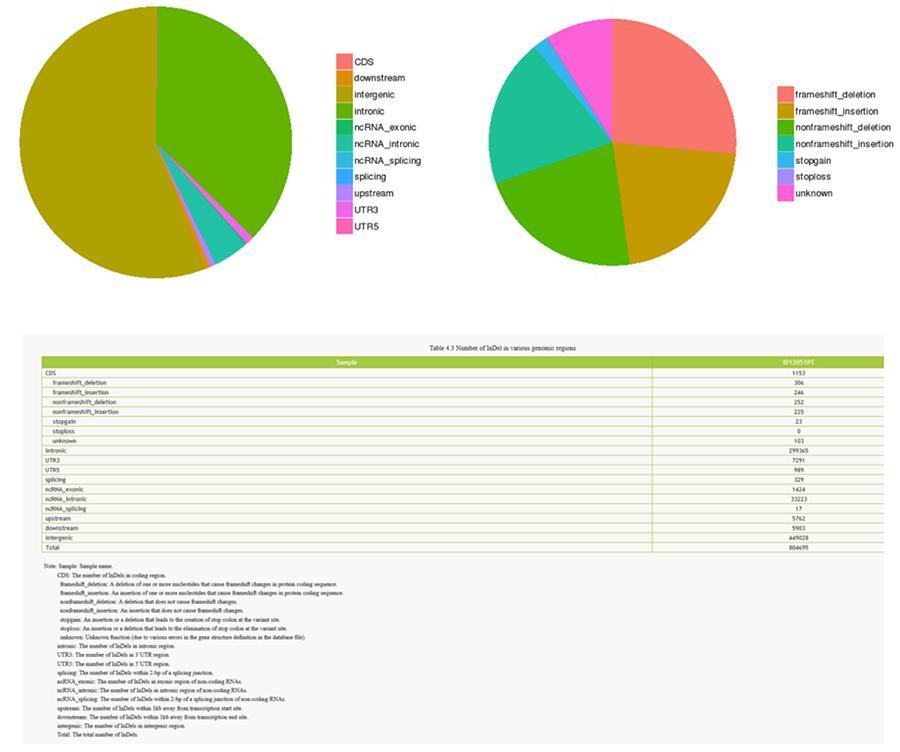
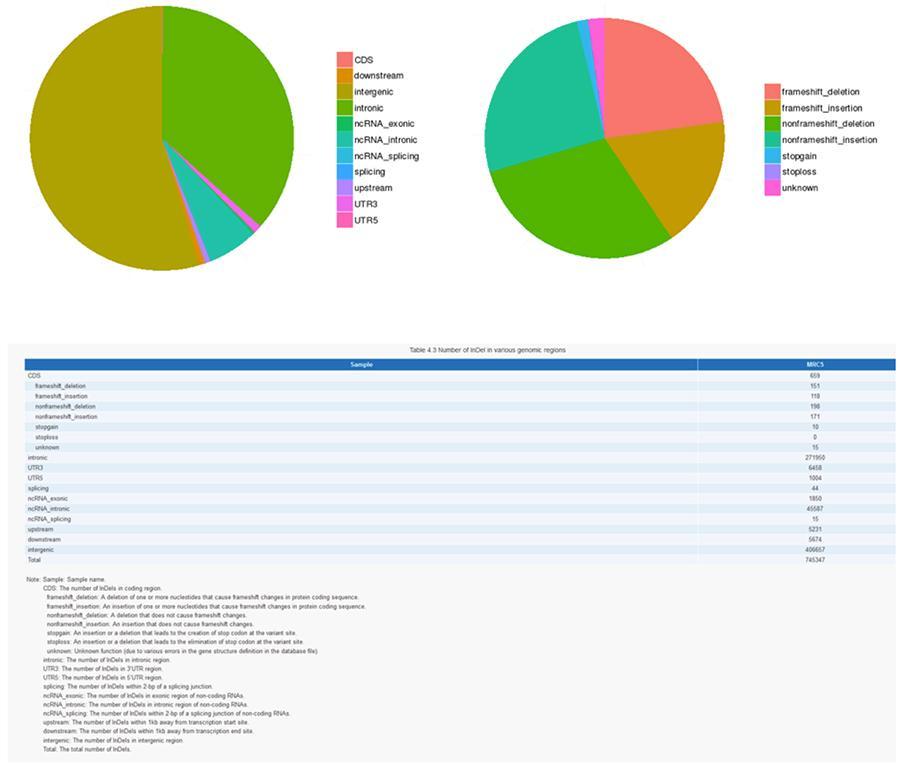
Les variantes de bases d'ADN uniques (SNP, prononcé «snip») sont des polymorphismes, c'est-à-dire des variations du matériel génétique, portées par un seul nucléotide. Les «InDels», d'autre part, sont de petites insertions et suppressions de moins de 50 pb de longueur et constituent une autre classe de variantes génomiques dans le génome humain.
SNP (variantes nucléotidiques simples) - ID12051PT
SNP (variantes nucléotidiques simples) - MRC-5
INDEL (petites insertions / suppressions) - ID12051PT
INDEL (petites insertions / suppressions) - MRC-5
CNV (Copy Number Variants) et SV (Structural Variants)
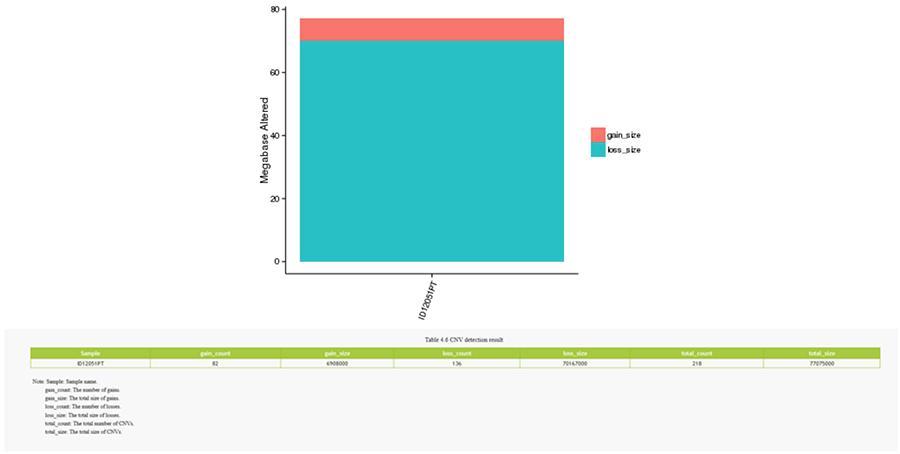
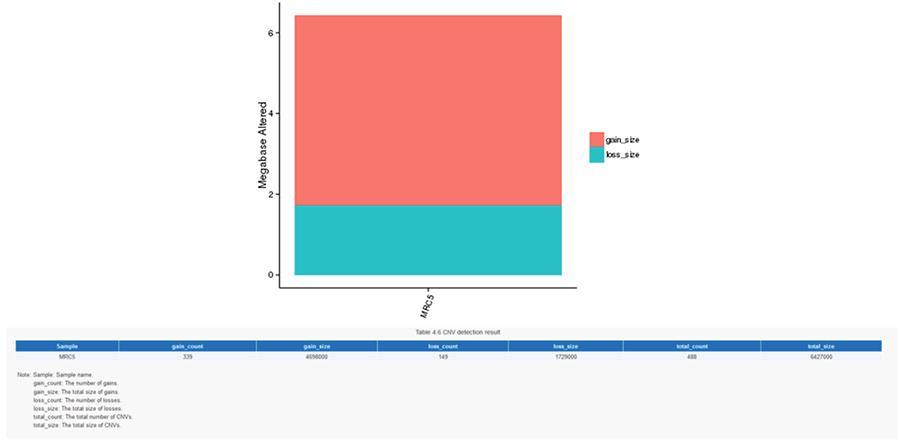
Les variantes du nombre de copies (CNV) sont des variantes génomiques en raison des variations du nombre de copies de fragments relativement gros (plus de 50 pb) entre les génomes individuels. Il existe deux types de CNV: le type «gain» (gain de copie) et le type «perte» (perte de copies).
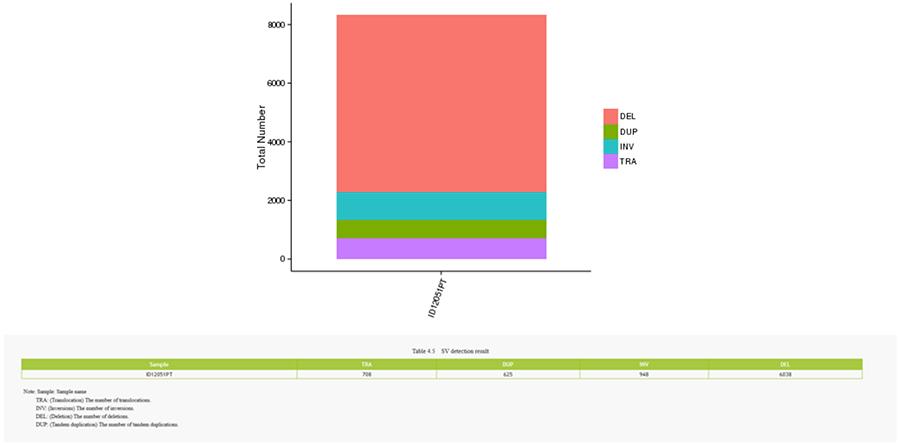
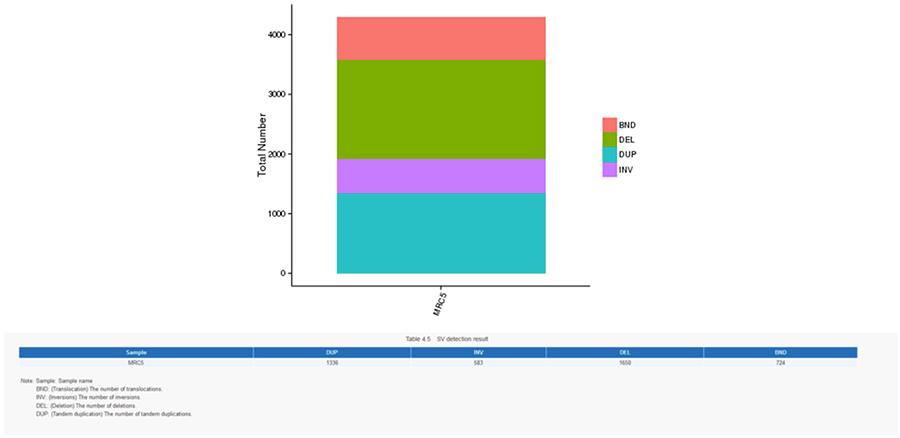
Les variantes structurelles (SV) sont des variantes génomiques avec des tailles relativement grandes (> 50 pb), y compris les délétions, les duplications, les insertions, les inversions et les translocations.
CNV (variations du nombre d'exemplaires)
SV (variantes structurelles)
Visualisation circulaire des génomes (intrigue circulaire)
Une représentation graphique des deux génomes appelés «intrigue circos» est présentée ci-dessous.
Signification des différents cercles concentriques
- Le cercle extérieur (le premier cercle) est le nombre de chromosomes.
- Le deuxième anneau (bleu) représente la couverture des lectures de style histogramme. Chaque histogramme est la couverture moyenne d'une zone de 0,5 Mbp.
- Le troisième anneau (noir) représente la densité des INDEL dans un style de «graphique de dispersion». Chaque point noir est calculé comme le nombre d'INDEL (petites insertions / suppressions) dans une plage de 1 Mbp.
- Le quatrième anneau (vert) représente la densité snp dans un style de «graphique de dispersion». Chaque point vert est calculé comme le nombre de SNP (variantes nucléotidiques simples) dans une plage de 1 Mbp.
- Le cinquième anneau représente la proportion d'homozygotie (orange) et d'hétérozygotie (gris) SNP dans le style histogramme. Chaque histogramme est calculé à partir d'une région de 1 Mbp.
- Le sixième anneau représente l'inférence de CNV (Variantes en nombre d'exemplaires). Le rouge signifie gagner des morceaux d'ADN et le vert signifie perdre.
- L'anneau le plus central représente l'inférence des SV (variantes structurelles) dans les régions exoniques et d'épissage. ENTRE (orange, translocations), INS (vert, insertions), DEL (suppressions, gris), DUP (duplications, rose) et INV (inversions, bleu).
Conclusions
Les deux génomes présentent des différences importantes. En particulier, le génome du vaccin se révèle fortement modifié par rapport à celui de la lignée cellulaire déposée en 1966. Il faut dire que nous n'avons pas eu la possibilité de séquencer la lignée cellulaire utilisée par GlaxoSmithKline pour la production du vaccin Priorix Tetra, mais celle du stock d'origine. Les sociétés pharmaceutiques qui utilisent des lignées cellulaires pour la production de médicaments et de vaccins ont leur lignée MRC-5 mais cela ne change pas nos soupçons, c'est le fait que le contrôle de la stabilité génétique n'est pas requis périodiquement et c'est très grave à notre avis , surtout parce qu'il est permis d'être présent sans limite.

