Comparación entre el genoma humano contenido en Priorix Tetra y en la línea celular MRC-5

Breve presentación de los resultados.
Presentamos hoy otro estudio muy importante sobre la secuenciación genómica de la línea celular que se encuentra dentro de la vacuna cuadrivalente MPRV (sarampión, rubéola, paperas y varicela) Priorix Tetra, y el de la línea celular MRC-5 (que es la que se utiliza en el desarrollo de la vacuna). vacuna en sí).
Resumiendo los informes publicados anteriores:
- Primero demostramos cómo la cantidad de ADN presente en la vacuna mencionada anteriormente estaba muy por encima de lo permitido;
- luego verificamos que la línea celular es en realidad el declarado MRC-5;
- posteriormente procedimos a secuenciación del genoma de la línea celular extraída directamente de la vacuna, destacando mutaciones muy pesadas que conllevan la posibilidad de riesgos graves para la salud en el uso de esta vacuna.
Lo que presentamos aquí es precisamente el trabajo de comparación entre los dos genomas: lo que realmente encontramos en la vacuna y el del MRC-5 comprado en la base de datos ATCC.
El resultado es que las dos líneas presentan diferencias importantes desde el punto de vista de la estabilidad genética. En particular, el genoma de la vacuna parece estar muy modificado en comparación con el de la línea celular depositada en 1966.
Esto significa que los productores, en este caso del Priorix tetra, han comprado la línea celular en condiciones óptimas desde el punto de vista de la estabilidad genética y con el uso continuo a lo largo del tiempo para la producción, han puesto en el mercado vacunas que contienen material genético humano progresivamente cada vez más modificado y peligroso para la salud de los vacunados.
Dado que las agencias reguladoras no requieren un control periódico de la estabilidad genética de las líneas fetales y ni siquiera establecen un umbral mínimo de seguridad, podemos deducir que es un requisito fundamental para la calidad y seguridad de las vacunas que han sido obtenido a partir de líneas celulares humanas, es decir, la necesidad de verificar que no adquieren mutaciones y reordenamientos potencialmente peligrosos y garantizar la eliminación de residuos de procesamiento de material genético si están presentes.
No nos corresponde a nosotros identificar qué parte del proceso de producción o conservación puede afectar la calidad y la seguridad del producto, pero lo que podemos testificar hoy al publicar este informe es que es urgente que los organismos de control realicen un examen exhaustivo de los resultados que nuestros análisis han destacado. en estos dos años, centrando la atención en el producto terminado, vendido y administrado y no solo en los "ingredientes" individuales autorizados.
Recuerde una vez más que estos productos forman parte del calendario de vacunación italiano y actualmente se administran obligatoriamente a la población pediátrica de nuestro país, así como a otros países europeos y en general en todo el mundo.
Los organismos de control tienen una enorme responsabilidad por la seguridad, que actualmente se anuncia con palabras, pero que, de hecho, aparentemente, y como se desprende de nuestros análisis, no se puede garantizar y, de hecho, se ignora claramente.
Visualización circular de genomas (trama circos)
A continuación se muestra una representación gráfica de los dos genomas llamada 'trama de circos'.
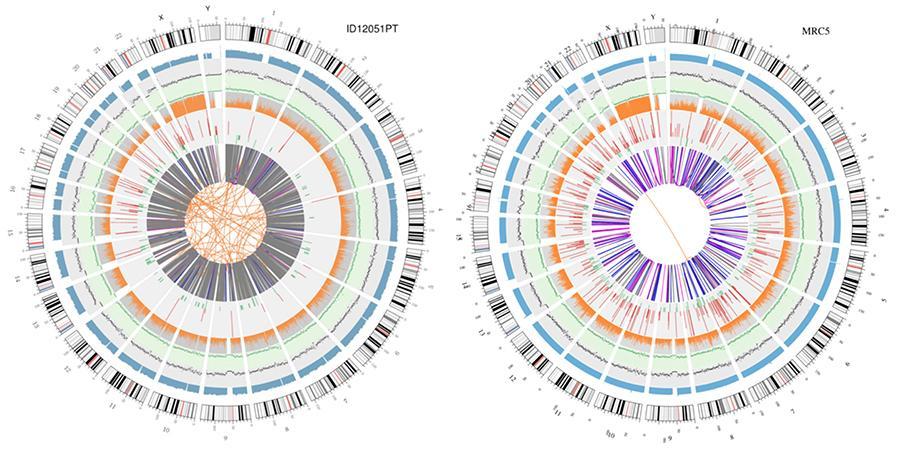
Significado de los diversos círculos concéntricos.
- El círculo externo (el primer círculo) es el número de cromosoma.
- El segundo anillo (azul) representa la cobertura de las lecturas de estilo de histograma. Cada histograma es la cobertura promedio de un área de 0,5 Mbp.
- El tercer anillo (negro) representa la densidad de los INDEL en un estilo de "gráfico de dispersión". Cada punto negro se calcula como el número de INDEL (pequeñas inserciones / eliminaciones) en un rango de 1 Mbp.
- El cuarto anillo (verde) representa la densidad snp en un estilo de 'gráfico de dispersión'. Cada punto verde se calcula como el número de SNP (variantes de un solo nucleótido) en un rango de 1 Mbp.
- El quinto anillo representa la proporción de SNP de homocigosidad (naranja) y heterocigosidad (gris) en el estilo de histograma. Cada histograma se calcula a partir de una región de 1 Mbp.
- El sexto anillo representa la inferencia de CNV (Variantes en número de copias). Rojo significa ganar pedazos de ADN y verde significa pérdida.
- El anillo más central representa la inferencia de SV (variantes estructurales) en las regiones exónicas y de empalme. ENTRE (naranja, translocaciones), INS (verde, inserciones), DEL (eliminaciones, gris), DUP (duplicaciones, rosa) e INV (inversiones, azul).
Conclusiones
Los dos genomas muestran diferencias importantes. En particular, el genoma de la vacuna se ve muy modificado en comparación con el de la línea celular depositada en 1966. Hay que decir que no tuvimos la posibilidad de secuenciar la línea celular utilizada por GlaxoSmithKline para la producción de la vacuna Priorix Tetra, sino la del stock originales. Las compañías farmacéuticas que usan líneas celulares para la producción de medicamentos y vacunas tienen su línea MRC-5, pero esto no cambia nuestras sospechas, es el hecho de que el control de la estabilidad genética no es necesario periódicamente y esto es muy grave en nuestra opinión. , sobre todo porque se le permite estar presente sin límite.
Descargar: CORVELVA-comparación de humano genoma-Priorix-tetra-y-line-MRC-5.pdf
Comparación entre el genoma humano contenido en Priorix Tetra y en la línea celular MRC-5 (estándar ATCC MRC-5 ATCC® CCL-171 ™)
La presencia del virus de la rubéola se demostró secuenciando una biblioteca de secuencias de ARN extremadamente profunda (se produjeron aproximadamente 260 millones de secuencias de Illumina). Se detectaron 114 de 260 millones de secuencias, lo que equivale al 0.00004% de la secuencia total. Las secuencias del genoma de la rubéola se confirmaron manualmente con el software BLAST (Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov/Blast.cgi). La biblioteca secuenciada a una profundidad inferior (aproximadamente 12 millones de secuencias emparejadas Illumina, equivalentes a 6 millones de fragmentos de biblioteca secuenciada) no había detectado la presencia de NINGUNA lectura atribuible a la rubéola en este lote.
Las secuencias de los otros virus presentes en la vacuna (varicela, sarampión y paperas) también se validaron de la misma manera manual, lo que confirma que se asignaron correctamente.
Introducción
Los secuenciadores de nueva generación se han convertido en instrumentos de elección para análisis en profundidad en el campo de la biología y la medicina, especialmente el de precisión. Estas herramientas permiten un enfoque nuevo y más global de una serie de aplicaciones, como la secuenciación de novo, la metagenómica, la epigenómica, la secuenciación del transcriptoma y la secuenciación del genoma.
Esta última aplicación (re-secuenciación) está muy extendida en el campo humano tanto para fines de investigación como de diagnóstico y consiste en la secuenciación con tecnología NGS (secuenciación de próxima generación) de un genoma individual completo para mapear mutaciones de nucleótidos individuales (SNP, recorte de pronunciación) '), se produjeron inserciones y deleciones de secuencias más o menos largas en ciertas posiciones del genoma y variaciones en el número de copias de porciones de genoma / genes (CNV, variantes de número de copia).
El procedimiento de secuenciación requiere que el ADN de un individuo se rompa mecánicamente en pequeños fragmentos (400-500 pares de bases) y fragmentos de tractos de ADN artificiales llamados adaptadores estén unidos a los fragmentos, lo que permite unir los fragmentos de ADN humano a una superficie de vidrio sobre la cual se leen las bases (A, C, G, T). Las bases de ADN se leen incorporando reacciones químicas de nucleótidos marcados con moléculas fluorescentes. Los millones de secuencias (lecturas) obtenidas de la secuenciación en la superficie del vidrio se mapean en el genoma de referencia humano con el software apropiado y luego se identifican todas las variantes presentes en el genoma analizado en comparación con la referencia.
Este mismo procedimiento se realizó en el genoma humano presente en el lote Priorix Tetra. n. A71CB256A y en el ADN extraído de la línea celular MRC-5, depositado en 1966 en ATCC (MRC-5 ATCC® CCL-171 ™).
ATCC es la organización líder mundial de recursos y estándares de materiales biológicos cuya misión se centra en la adquisición, autenticación, producción, almacenamiento, desarrollo y distribución de microorganismos de referencia, líneas celulares y otros materiales biológicos estándar. Fue establecido en 1925, cuando un comité de científicos reconoció la necesidad de una colección centralizada de materiales biológicos que pudieran servir a los científicos de todo el mundo.
La línea celular MRC-5 se deriva del tejido pulmonar normal de un feto masculino de 14 semanas de edad y fue depositada en ATCC por JP Jacobs en septiembre de 1966. Se dice que estas células pueden duplicar de 42 a 46 poblaciones antes del inicio. de senescencia El artículo de referencia para esta línea celular se remonta a 1970: https://www.nature.com/articles/227168a0
Resultados
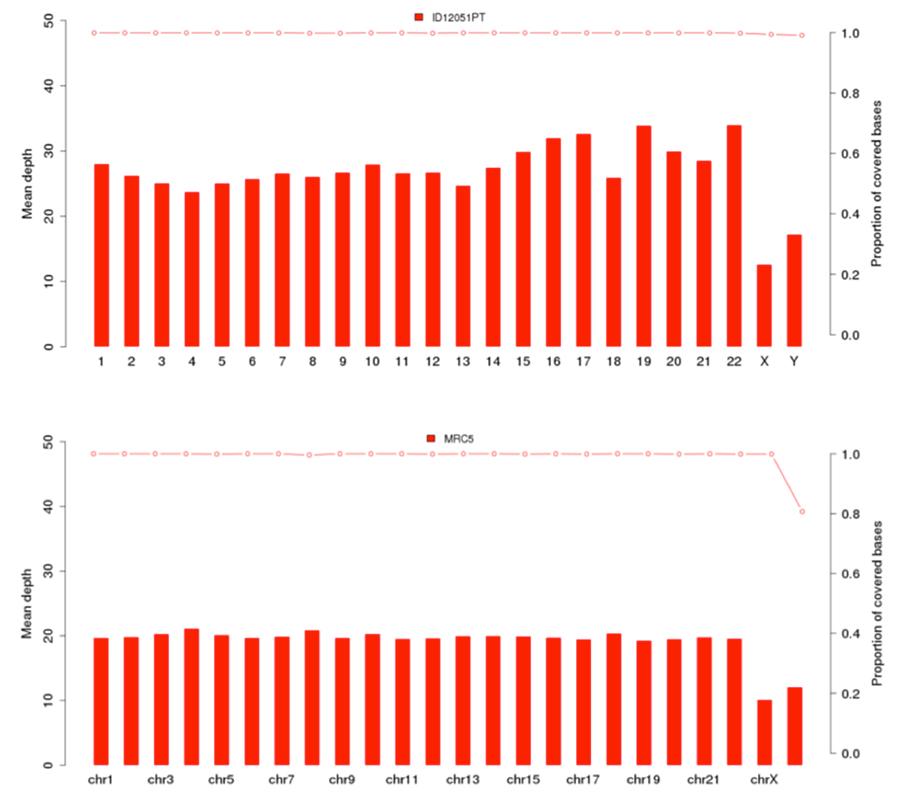
Como se mostró anteriormente, el ADN humano presente en el lote Priorix Tetra. n. A71CB256A (indicado en los siguientes gráficos como muestra ID12051PT) es un genoma individual completo o ADN genómico de todos los cromosomas de un individuo masculino presente. Los gráficos de cobertura (profundidad media = cobertura promedio) de los 22 cromosomas humanos y de los cromosomas X e Y se muestran a continuación, para la vacuna (imagen de arriba) y la línea celular MRC5 (imagen de abajo).
Los resultados del análisis de los diversos tipos de variantes se muestran a continuación.
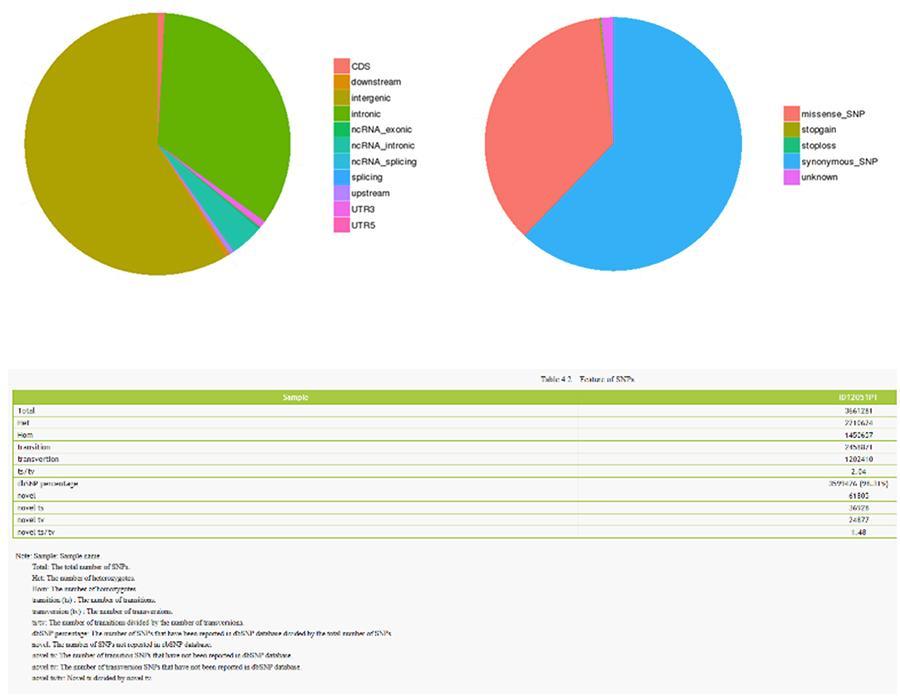
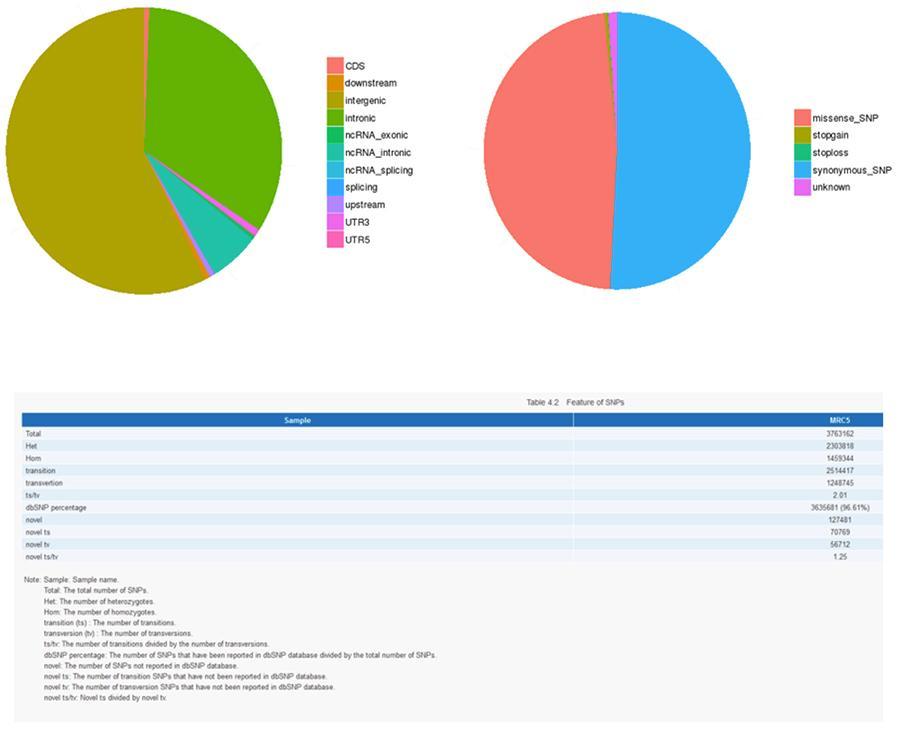
Variantes de un solo nucleótido (SNP) e inserciones / deleciones cortas (InDels)
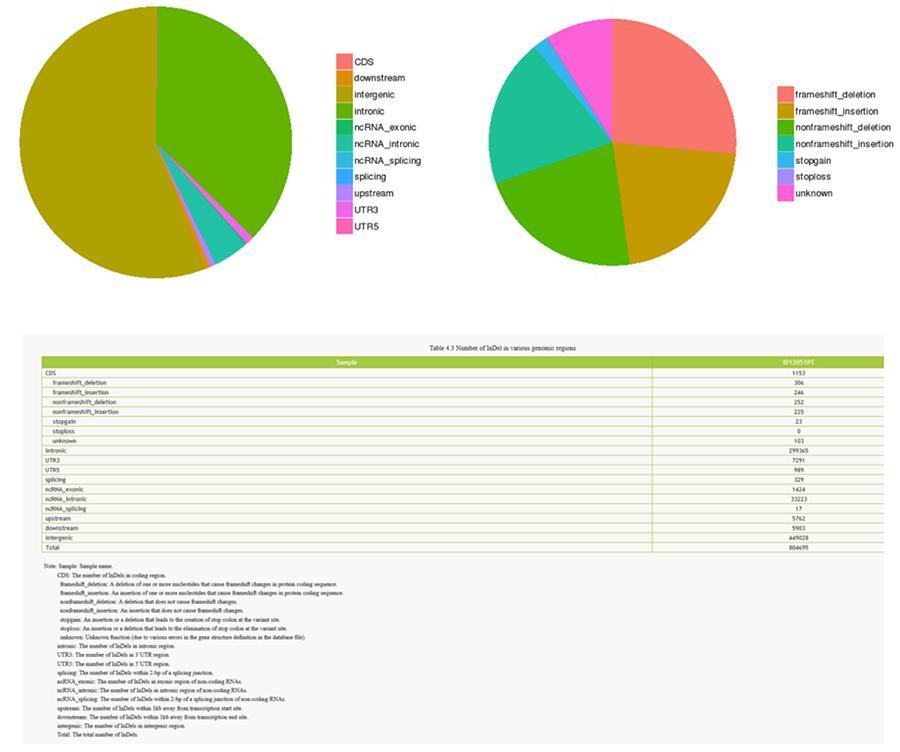
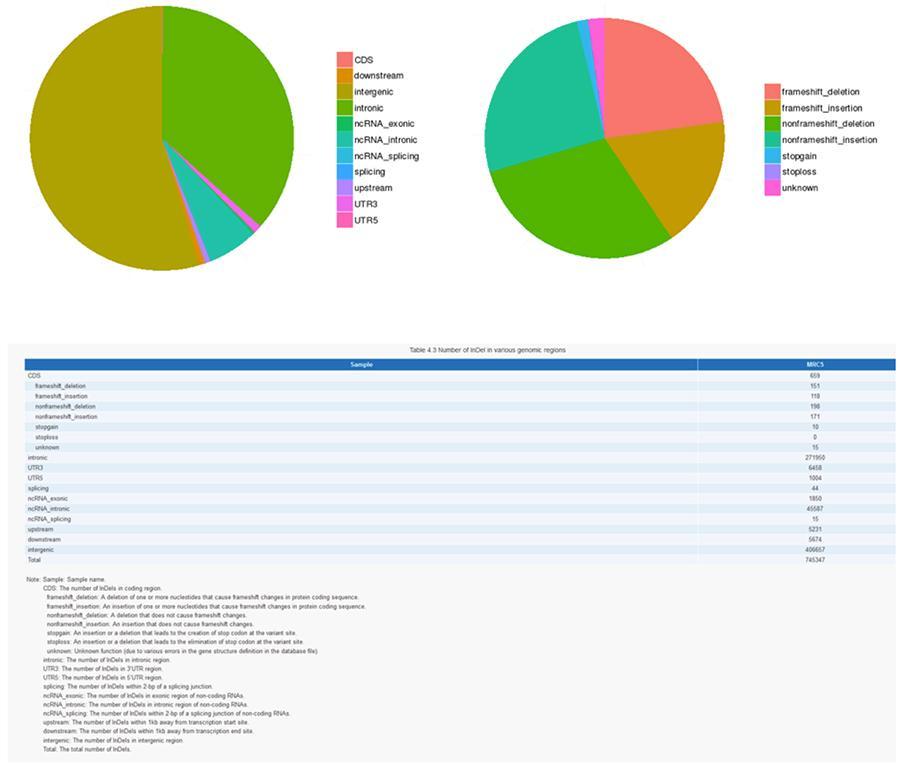
Las variantes de bases de ADN individuales (SNP, pronunciadas 'snip') son polimorfismos, es decir, variaciones del material genético, llevadas por un solo nucleótido. Los 'InDels', por otro lado, son pequeñas inserciones y deleciones de menos de 50 pb de longitud y constituyen otra clase de variantes genómicas en el genoma humano.
SNP (variantes de un solo nucleótido) - ID12051PT
SNP (variantes de un solo nucleótido) - MRC-5
INDEL (pequeñas inserciones / eliminaciones) - ID12051PT
INDEL (pequeñas inserciones / eliminaciones) - MRC-5
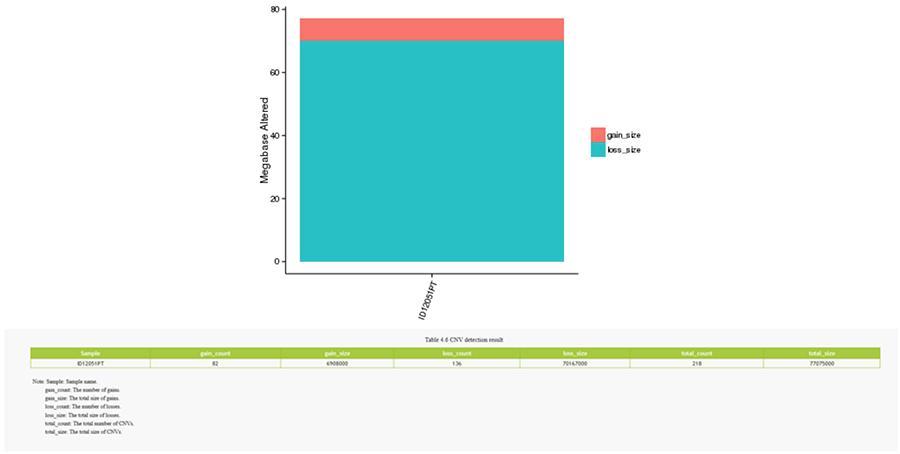
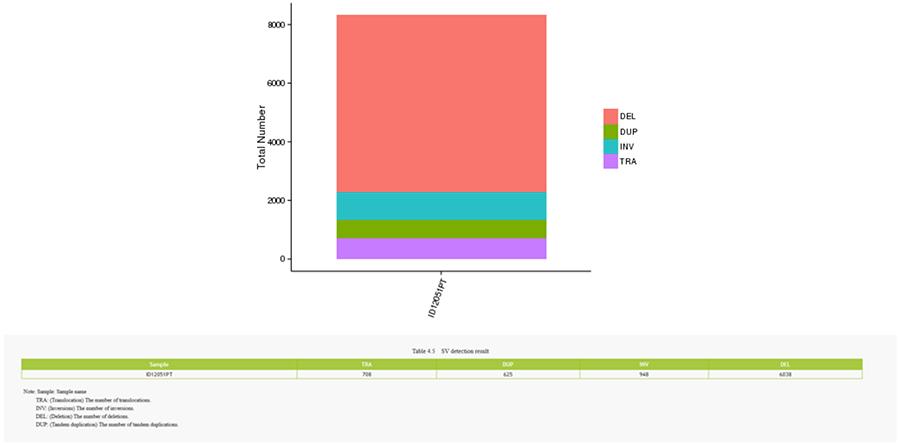
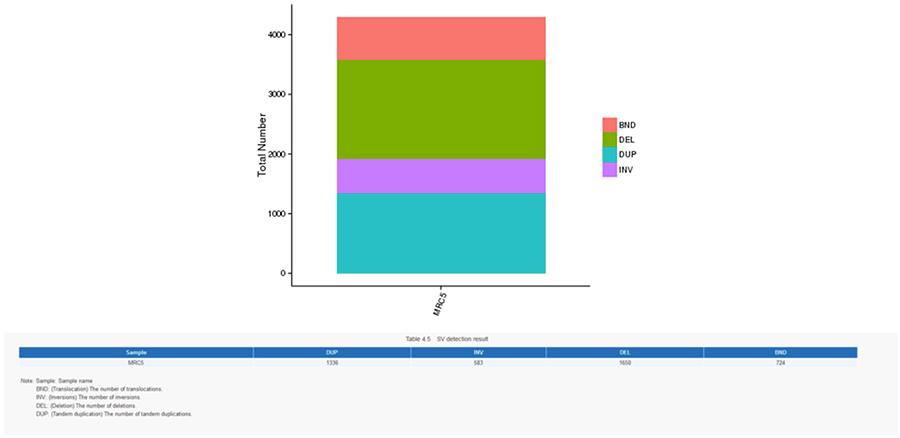
CNV (Variantes de número de copia) y SV (Variantes estructurales)
Las variantes en el número de copias (CNV) son variantes genómicas debido a las variaciones en el número de copias de fragmentos relativamente grandes (más de 50 pb) entre genomas individuales. Hay dos tipos de CNV: tipo 'ganancia' (ganancia de copia) y tipo 'pérdida' (pérdida de copias).
Las variantes estructurales (SV) son variantes genómicas con tamaños relativamente grandes (> 50 pb), que incluyen deleciones, duplicaciones, inserciones, inversiones y translocaciones.
CNV (variaciones en el número de copias)
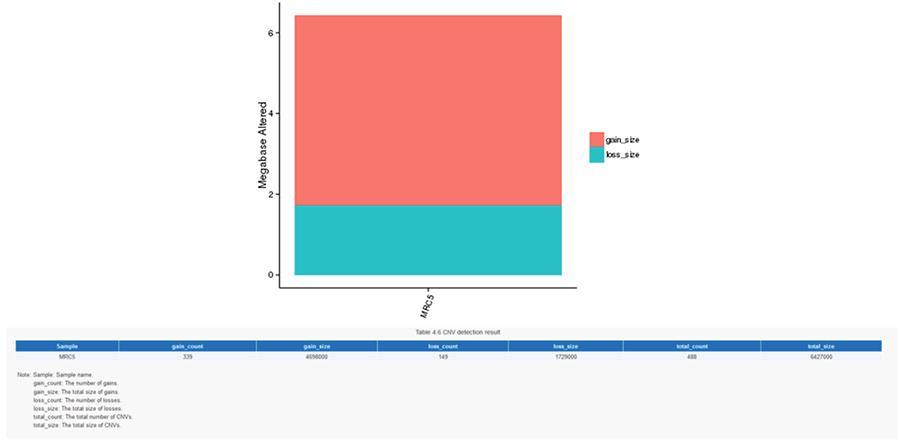
SV (variantes estructurales)
Visualización circular de genomas (trama circos)
A continuación se muestra una representación gráfica de los dos genomas llamada 'trama de circos'.
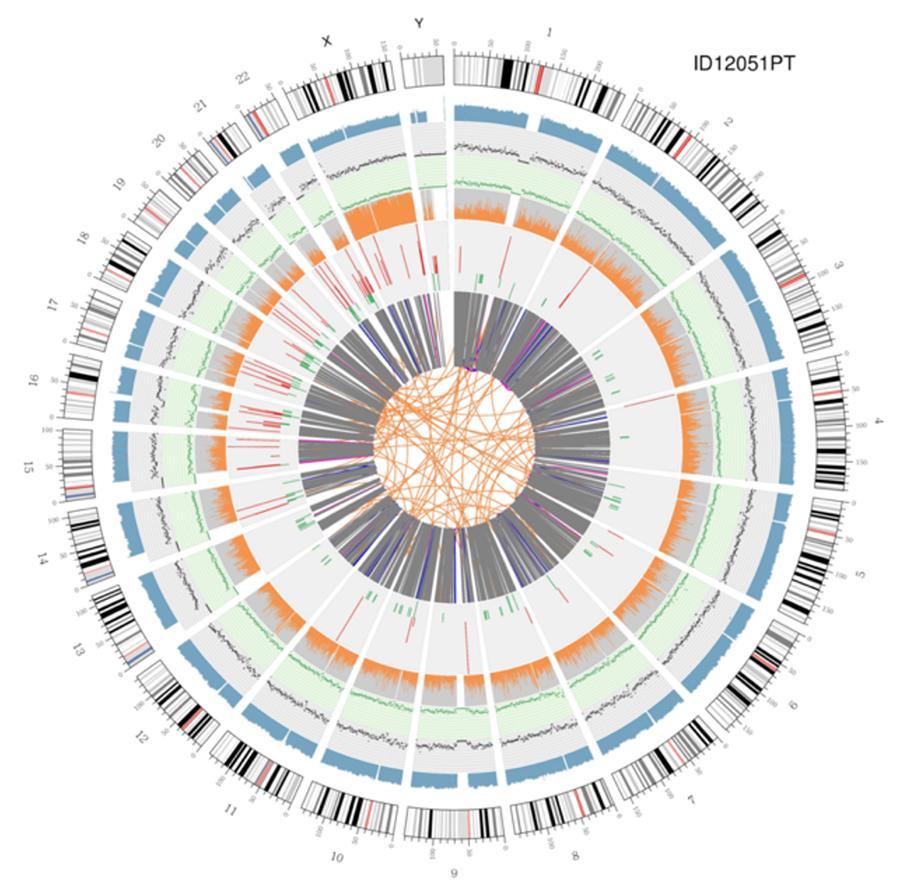
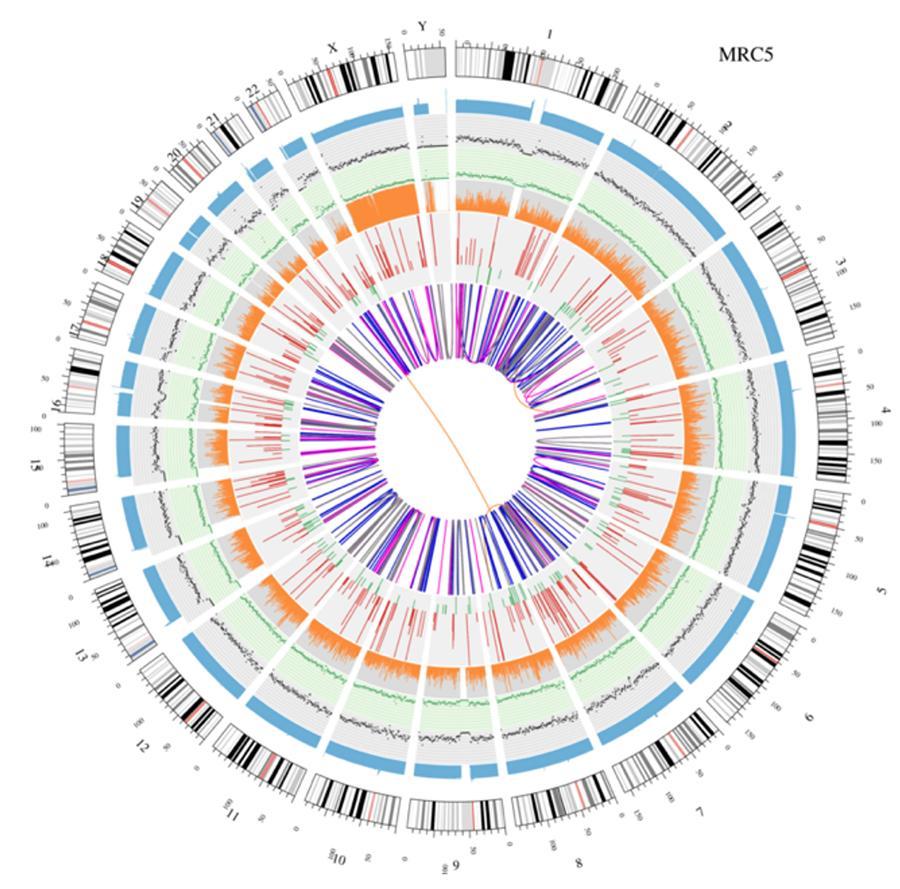
Significado de los diversos círculos concéntricos.
- El círculo externo (el primer círculo) es el número de cromosoma.
- El segundo anillo (azul) representa la cobertura de las lecturas de estilo de histograma. Cada histograma es la cobertura promedio de un área de 0,5 Mbp.
- El tercer anillo (negro) representa la densidad de los INDEL en un estilo de "gráfico de dispersión". Cada punto negro se calcula como el número de INDEL (pequeñas inserciones / eliminaciones) en un rango de 1 Mbp.
- El cuarto anillo (verde) representa la densidad snp en un estilo de 'gráfico de dispersión'. Cada punto verde se calcula como el número de SNP (variantes de un solo nucleótido) en un rango de 1 Mbp.
- El quinto anillo representa la proporción de SNP de homocigosidad (naranja) y heterocigosidad (gris) en el estilo de histograma. Cada histograma se calcula a partir de una región de 1 Mbp.
- El sexto anillo representa la inferencia de CNV (Variantes en número de copias). Rojo significa ganar pedazos de ADN y verde significa pérdida.
- El anillo más central representa la inferencia de SV (variantes estructurales) en las regiones exónicas y de empalme. ENTRE (naranja, translocaciones), INS (verde, inserciones), DEL (eliminaciones, gris), DUP (duplicaciones, rosa) e INV (inversiones, azul).
Conclusiones
Los dos genomas muestran diferencias importantes. En particular, el genoma de la vacuna se ve muy modificado en comparación con el de la línea celular depositada en 1966. Hay que decir que no tuvimos la posibilidad de secuenciar la línea celular utilizada por GlaxoSmithKline para la producción de la vacuna Priorix Tetra, sino la del stock originales. Las compañías farmacéuticas que usan líneas celulares para la producción de medicamentos y vacunas tienen su línea MRC-5, pero esto no cambia nuestras sospechas, es el hecho de que el control de la estabilidad genética no es necesario periódicamente y esto es muy grave en nuestra opinión. , sobre todo porque se le permite estar presente sin límite.

