Vergleich zwischen dem in Priorix Tetra und in der MRC-5-Zelllinie enthaltenen menschlichen Genom

Kurze Präsentation der Ergebnisse
Wir präsentieren heute eine weitere sehr wichtige Studie zur genomischen Sequenzierung der Zelllinie, die im vierwertigen Impfstoff MPRV (Masern, Röteln, Mumps und Windpocken) Priorix Tetra gefunden wurde, und der MRC-5-Zelllinie (die bei der Entwicklung der Impfstoff selbst).
Zusammenfassung der zuvor veröffentlichten Berichte:
- Wir haben zuerst gezeigt, wie viel DNA im oben genannten Impfstoff vorhanden ist war weit über dem erlaubten;
- Wir haben dann überprüft, ob die Zelllinie ist eigentlich das deklarierte MRC-5;
- wir gingen weiter zu Genomsequenzierung der direkt aus dem Impfstoff extrahierten ZelllinieHervorheben sehr schwerer Mutationen, die die Möglichkeit schwerwiegender Gesundheitsrisiken bei der Verwendung dieses Impfstoffs mit sich bringen.
Was wir hier präsentieren, ist genau die Vergleichsarbeit zwischen den beiden Genomen: Was wir wirklich im Impfstoff gefunden haben und das des MRC-5, das aus der ATCC-Datenbank gekauft wurde.
Das Ergebnis ist, dass die beiden Linien wichtige Unterschiede im Hinblick auf die genetische Stabilität aufweisen. Insbesondere scheint das Impfstoffgenom im Vergleich zu der 1966 abgelagerten Zelllinie stark verändert zu sein.
Dies bedeutet, dass die Hersteller, in diesem Fall des Priorix tetra, die Zelllinie unter optimalen Bedingungen unter dem Gesichtspunkt der genetischen Stabilität gekauft und bei kontinuierlicher Verwendung im Laufe der Zeit zum Zwecke der Herstellung schrittweise Impfstoffe auf den Markt gebracht haben, die menschliches genetisches Material enthalten zunehmend modifiziert und gefährlich für die Gesundheit der Geimpften.
Da die Aufsichtsbehörden keine regelmäßige Kontrolle der genetischen Stabilität fetaler Linien verlangen und nicht einmal eine Mindestsicherheitsschwelle festlegen, können wir daraus eine grundlegende Anforderung für die Qualität und Sicherheit der bisherigen Impfstoffe ableiten aus menschlichen Zelllinien gewonnen, dh die Notwendigkeit, zu überprüfen, ob sie keine potenziell gefährlichen Mutationen und Umlagerungen annehmen, und die Eliminierung von Verarbeitungsresten von genetischem Material zu gewährleisten, falls vorhanden.
Es ist nicht unsere Aufgabe, festzustellen, welcher Teil des Produktions- oder Konservierungsprozesses die Qualität und Sicherheit des Produkts beeinträchtigen kann. Was wir heute durch die Veröffentlichung dieses Berichts bezeugen können, ist, dass die Kontrollstellen dringend eine gründliche Prüfung der Ergebnisse durchführen müssen, die unsere Analysen hervorgehoben haben. In diesen zwei Jahren wurde die Aufmerksamkeit auf das fertige Produkt gerichtet, das verkauft und verabreicht wurde, und nicht nur auf die einzelnen zugelassenen "Inhaltsstoffe".
Denken Sie noch einmal daran, dass diese Produkte Teil des italienischen Impfkalenders sind und derzeit der pädiatrischen Bevölkerung unseres Landes sowie anderen europäischen Ländern und allgemein der ganzen Welt obligatorisch verabreicht werden.
Die Kontrollstellen haben eine enorme Verantwortung für die Sicherheit, die derzeit in Worten angekündigt wird, die jedoch offenbar und wie aus unseren Analysen hervorgeht, nicht garantiert werden kann und in der Tat eindeutig außer Acht gelassen wird.
Zirkuläre Visualisierung von Genomen (Circos Plot)
Eine grafische Darstellung der beiden Genome, die als "Circos Plot" bezeichnet wird, ist unten dargestellt.
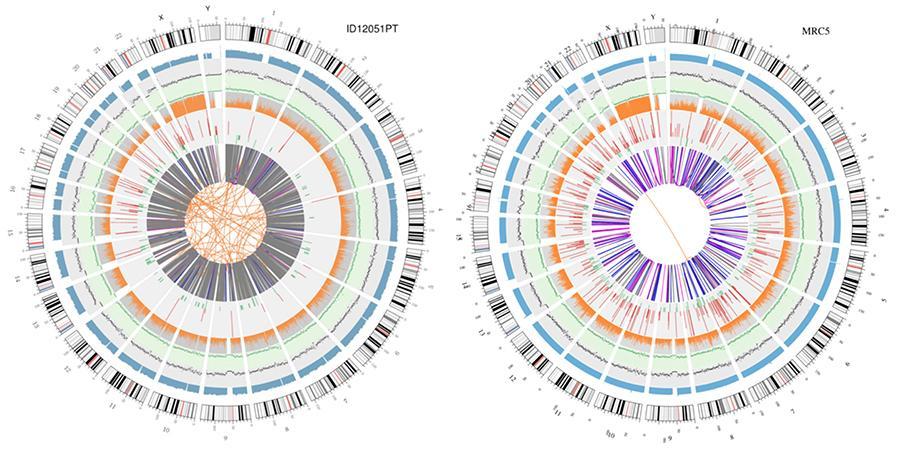
Bedeutung der verschiedenen konzentrischen Kreise
- Der äußere Kreis (der erste Kreis) ist die Chromosomenzahl.
- Der zweite Ring (blau) repräsentiert die Abdeckung von Lesevorgängen im Histogrammstil. Jedes Histogramm ist die durchschnittliche Abdeckung einer Fläche von 0,5 Mbit / s.
- Der dritte Ring (schwarz) repräsentiert die Dichte der INDELs in einem "Dispersionsgraphen" -Stil. Jeder Schwarzpunkt wird als Anzahl der INDELs (kleine Einfügungen / Löschungen) in einem Bereich von 1 Mbit / s berechnet.
- Der vierte Ring (grün) repräsentiert die Dichte snp in einem "Dispersionsgraphen" -Stil. Jeder grüne Punkt wird als Anzahl von SNPs (Einzelnukleotidvarianten) in einem Bereich von 1 Mbit / s berechnet.
- Der fünfte Ring repräsentiert den Anteil von Homozygotie (orange) und Heterozygotie (grau) SNP im Histogrammstil. Jedes Histogramm wird aus einer 1-Mbit / s-Region berechnet.
- Der sechste Ring repräsentiert die Inferenz von CNV (Varianten in Anzahl der Kopien). Rot bedeutet, DNA-Stücke zu gewinnen, und Grün bedeutet Verlust.
- Der zentralste Ring repräsentiert die Inferenz von SV (Strukturvarianten) in den exonischen und Spleißbereichen. ZWISCHEN (orange, Translokationen), INS (grün, Insertionen), DEL (Deletionen, grau), DUP (Duplikationen, pink) und INV (Inversionen, blau).
Schlussfolgerungen
Die beiden Genome zeigen wichtige Unterschiede. Insbesondere das Impfstoffgenom ist im Vergleich zu dem der 1966 hinterlegten Zelllinie stark verändert. Es muss gesagt werden, dass wir nicht die Möglichkeit hatten, die Zelllinie zu sequenzieren, die von GlaxoSmithKline für die Herstellung des Priorix Tetra-Impfstoffs verwendet wird, sondern die des Bestandes Original. Pharmaunternehmen, die Zelllinien zur Herstellung von Arzneimitteln und Impfstoffen verwenden, haben ihre MRC-5-Linie, aber dies ändert nichts an unserem Verdacht, dh der Tatsache, dass keine genetische Stabilitätskontrolle in regelmäßigen Abständen erforderlich ist, und dies ist unserer Meinung nach sehr ernst , vor allem, weil es unbegrenzt präsent sein darf.
Herunterladen: CORVELVA-Vergleich-Human-Genom-Priorix-Tetra-and-line-MRC-5.pdf
Vergleich zwischen dem in Priorix Tetra und in der MRC-5-Zelllinie enthaltenen menschlichen Genom (ATCC-Standard MRC-5 ATCC® CCL-171 ™)
Das Vorhandensein des Rötelnvirus wurde durch Sequenzierung einer extrem tiefen RNA-seq-Bibliothek (ungefähr 260 Millionen produzierte Illumina-Sequenzen) nachgewiesen. 114 von 260 Millionen Sequenzen wurden nachgewiesen, was 0.00004% der Gesamtsequenz entspricht. Die Rötelngenomsequenzen wurden dann manuell mit der BLAST-Software (Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov/Blast.cgi). Die sequenzierte Bibliothek in geringerer Tiefe (ungefähr 12 Millionen Illumina-gekoppelte Sequenzen, was 6 Millionen sequenzierten Bibliotheksfragmenten entspricht) hatte in dieser Charge keine ANY-Reads nachgewiesen, die auf Röteln zurückzuführen waren.
Die Sequenzen der anderen im Impfstoff vorhandenen Viren (Windpocken, Masern und Mumps) wurden ebenfalls auf die gleiche manuelle Weise validiert, um zu bestätigen, dass sie korrekt zugeordnet wurden.
Einführung
Die Sequenzer der neuen Generation sind zu Instrumenten der Wahl für eingehende Analysen auf dem Gebiet der Biologie und Medizin geworden, insbesondere im Bereich der Präzision. Diese Tools ermöglichen einen neuen und globaleren Ansatz für eine Reihe von Anwendungen wie De-novo-Sequenzierung, Metagenomik, Epigenomik, Transkriptomsequenzierung und Genom-Re-Sequenzierung.
Diese letztere Anwendung (Re-Sequenzierung) ist im menschlichen Bereich sowohl für Forschungs- als auch für diagnostische Zwecke sehr verbreitet und besteht in der Sequenzierung eines gesamten individuellen Genoms mit NGS-Technologie (Next Generation Sequencing), um einzelne Nukleotidmutationen (SNP, Aussprache-Snip) abzubilden '), Insertionen und Deletionen von mehr oder weniger langen Sequenzen traten an bestimmten Positionen des Genoms und Variationen in der Anzahl der Kopien von Teilen des Genoms / der Gene (CNV, Copy Number Variants) auf.
Das Sequenzierungsverfahren erfordert, dass die DNA eines Individuums mechanisch in kleine Fragmente (400-500 Basenpaare) zerlegt wird und Fragmente künstlicher DNA-Trakte, sogenannte Adapter, an die Fragmente gebunden werden, an die die Fragmente menschlicher DNA gebunden werden können eine Glasoberfläche, auf der dann die Basen abgelesen werden (A, C, G, T). Die DNA-Basen werden durch Einbau chemischer Reaktionen von mit fluoreszierenden Molekülen markierten Nukleotiden gelesen. Die Millionen von Sequenzen (Reads), die durch Sequenzierung auf der Glasoberfläche erhalten wurden, werden dann mit geeigneter Software auf das menschliche Referenzgenom abgebildet, und dann werden alle im analysierten Genom vorhandenen Varianten im Vergleich zur Referenz identifiziert.
Das gleiche Verfahren wurde an dem in der Priorix Tetra-Charge vorhandenen menschlichen Genom durchgeführt. n. A71CB256A und auf der DNA, die aus der MRC-5-Zelllinie extrahiert wurde und 1966 bei ATCC hinterlegt wurde (MRC-5 ATCC® CCL-171 ™).
ATCC ist die weltweit führende Organisation von Ressourcen und Standards für biologisches Material, deren Mission sich auf den Erwerb, die Authentifizierung, die Produktion, die Konservierung, die Entwicklung und den Vertrieb von Referenzmikroorganismen, Zelllinien und anderen biologischen Standardmaterialien konzentriert. Es wurde 1925 gegründet, als ein Wissenschaftskomitee die Notwendigkeit einer zentralisierten Sammlung biologischer Materialien erkannte, die Wissenschaftlern auf der ganzen Welt dienen könnten.
Die MRC-5-Zelllinie stammt aus normalem Lungengewebe eines 14 Wochen alten männlichen Fötus und wurde im September 1966 von JP Jacobs in ATCC hinterlegt. Diese Zellen sollen vor der Initiierung 42 bis 46 Populationen verdoppeln können. der Seneszenz. Der Referenzartikel für diese Zelllinie stammt aus dem Jahr 1970: https://www.nature.com/articles/227168a0
Ergebnisse
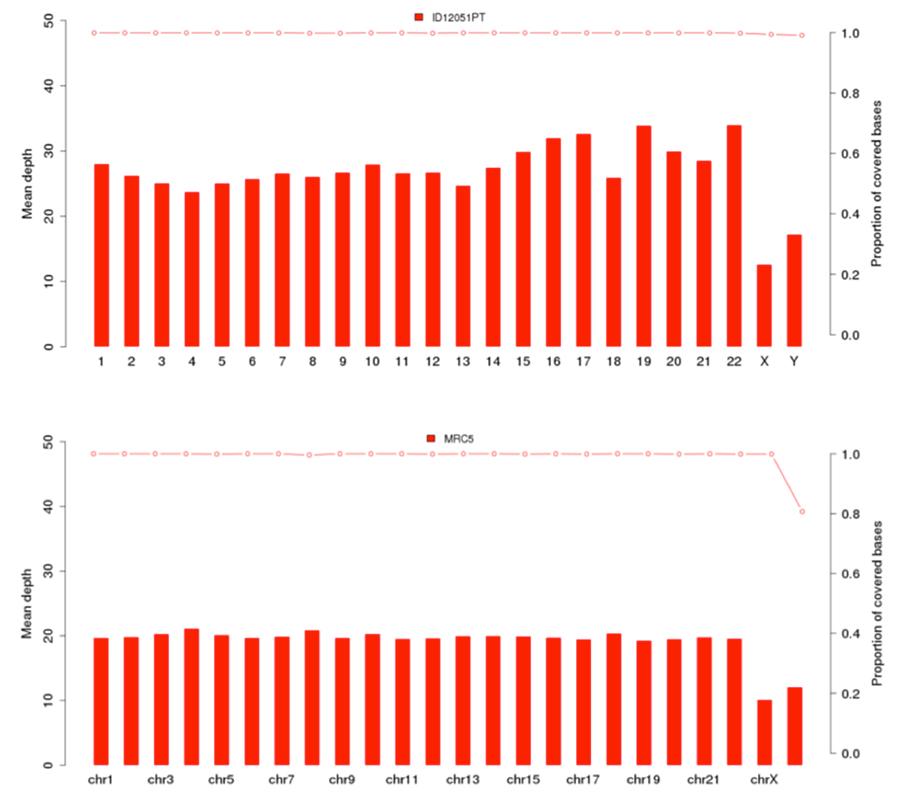
Wie zuvor gezeigt, ist die menschliche DNA in der Priorix Tetra-Charge vorhanden. n. A71CB256A (in den folgenden Diagrammen als Probe ID12051PT angegeben) ist ein vollständiges individuelles Genom oder genomische DNA aller Chromosomen eines männlichen Individuums ist vorhanden. Die Abdeckungsgraphen (mittlere Tiefe = durchschnittliche Abdeckung) der 22 menschlichen Chromosomen und der X- und Y-Chromosomen sind unten für den Impfstoff (Bild oben) und die MRC5-Zelllinie (Bild unten) gezeigt.
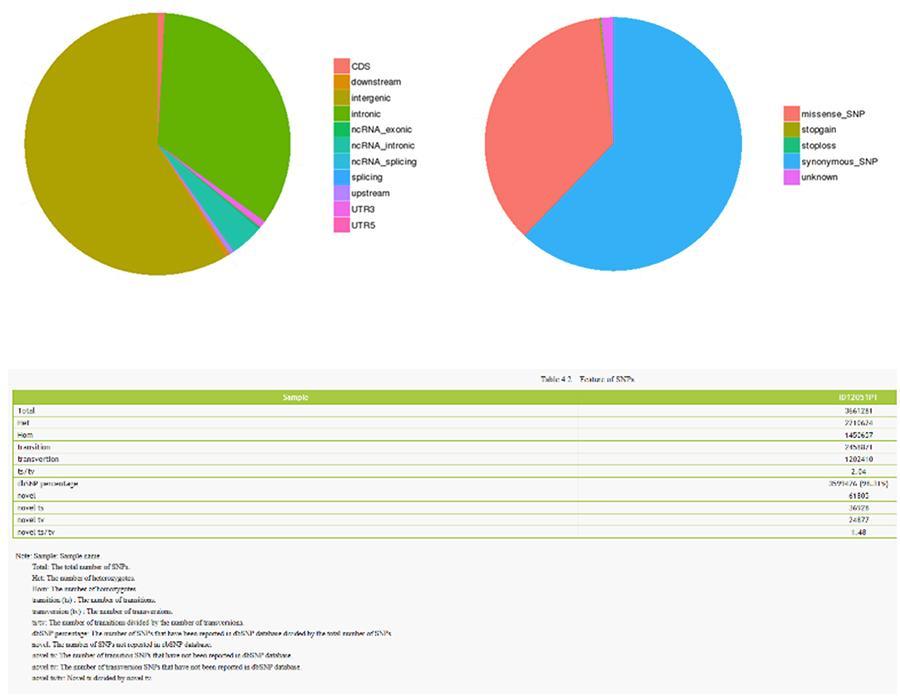
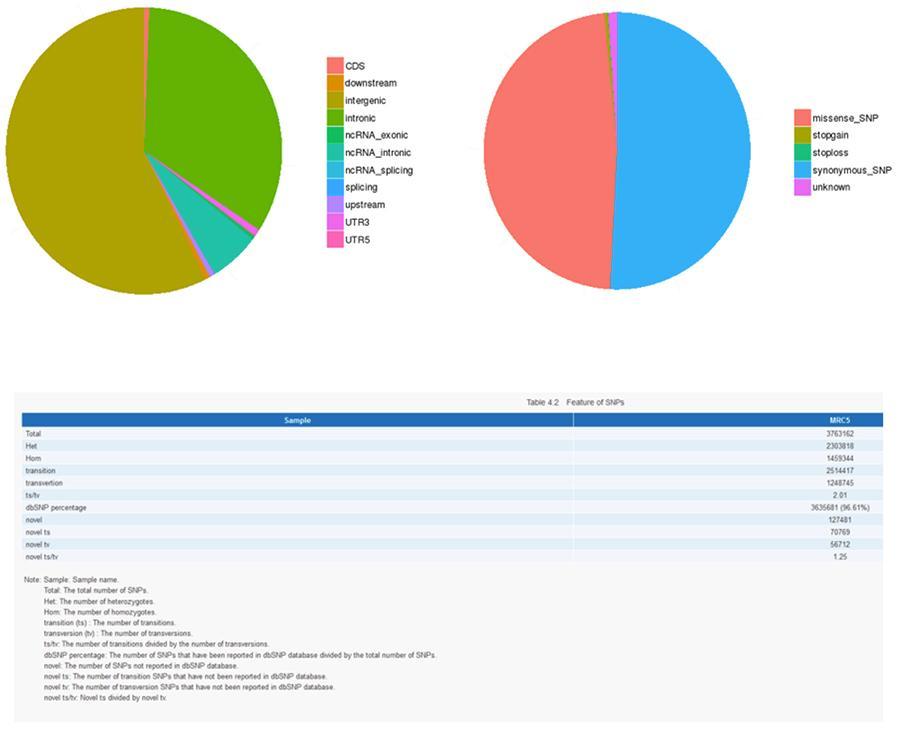
Die Ergebnisse der Analyse der verschiedenen Arten von Varianten sind unten gezeigt.
Einzelne Nukleotidvarianten (SNP) und kurze Insertionen / Deletionen (InDels)
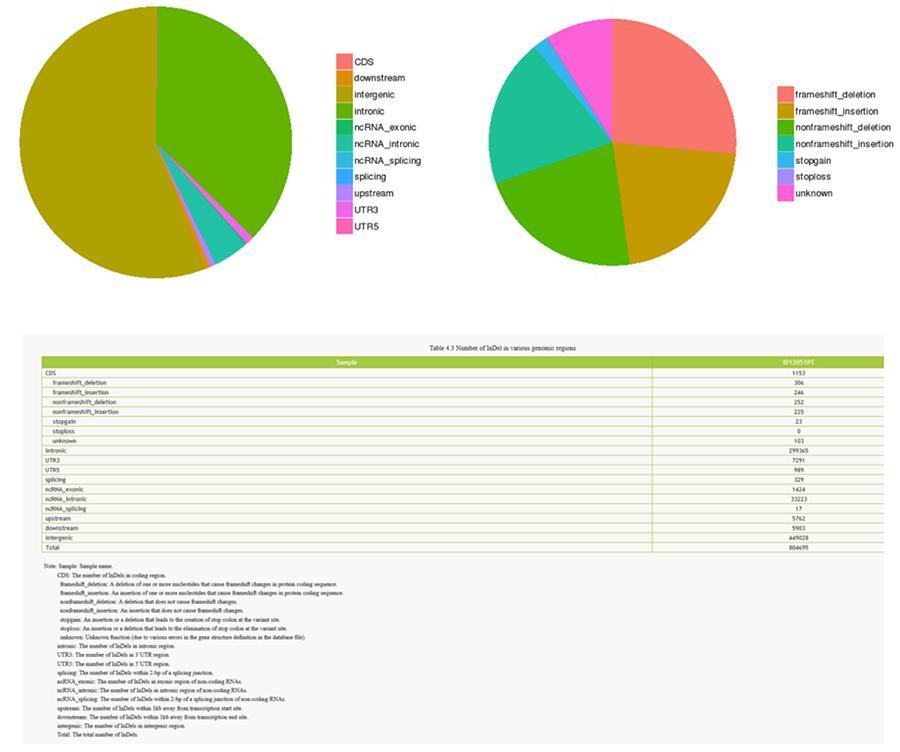
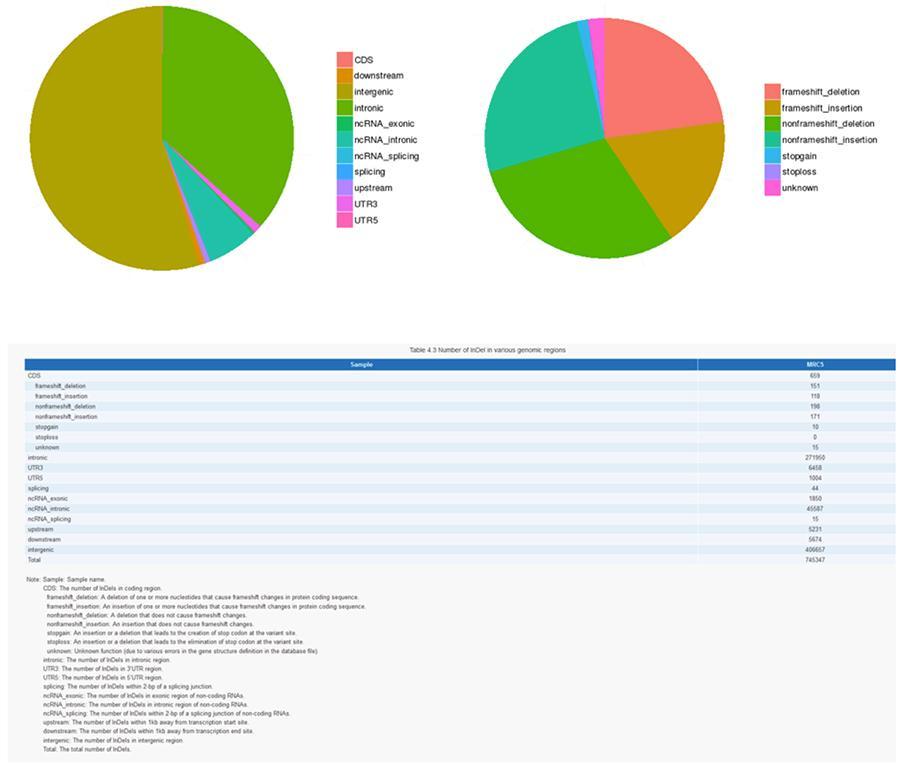
Die Varianten einzelner DNA-Basen (SNP, ausgesprochen "snip") sind Polymorphismen, dh Variationen des genetischen Materials, die von einem einzelnen Nukleotid getragen werden. Die 'InDels' hingegen sind kleine Insertionen und Deletionen mit einer Länge von weniger als 50 bp und bilden eine weitere Klasse genomischer Varianten im menschlichen Genom.
SNP (Einzelnukleotidvarianten) - ID12051PT
SNP (Einzelnukleotidvarianten) - MRC-5
INDELs (kleine Einfügungen / Löschungen) - ID12051PT
INDELs (kleine Einfügungen / Löschungen) - MRC-5
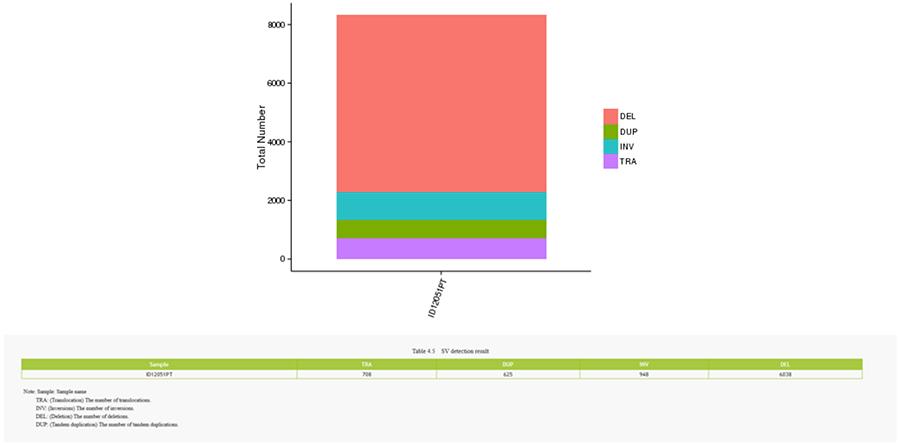
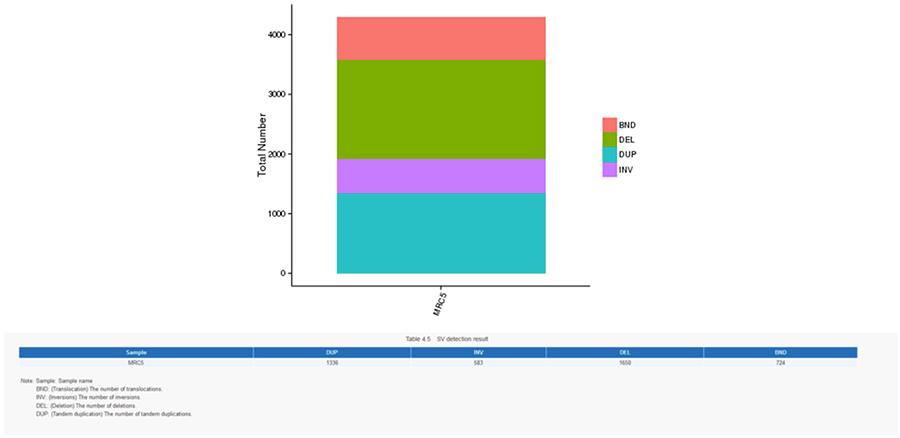
CNV (Copy Number Variants) und SV (Structural Variants)
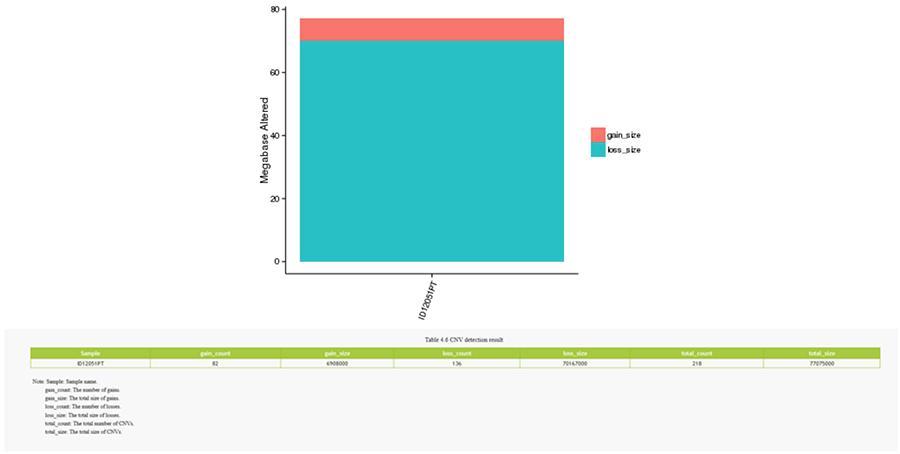
Varianten in der Anzahl der Kopien (CNV) sind genomische Varianten aufgrund von Variationen in der Anzahl der Kopien relativ großer Fragmente (länger als 50 bp) zwischen einzelnen Genomen. Es gibt zwei Arten von CNV: den Typ "Gewinn" (Kopiergewinn) und den Typ "Verlust" (Verlust von Kopien).
Strukturvarianten (SV) sind genomische Varianten mit relativ großen Größen (> 50 bp), einschließlich Deletionen, Duplikationen, Insertionen, Inversionen und Translokationen.
CNV (Variationen in der Anzahl der Kopien)
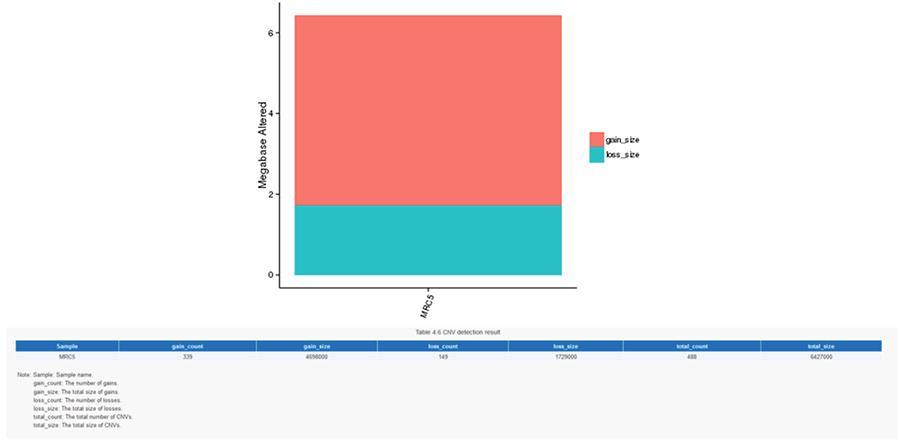
SV (Strukturvarianten)
Zirkuläre Visualisierung von Genomen (Circos Plot)
Eine grafische Darstellung der beiden Genome, die als "Circos Plot" bezeichnet wird, ist unten dargestellt.
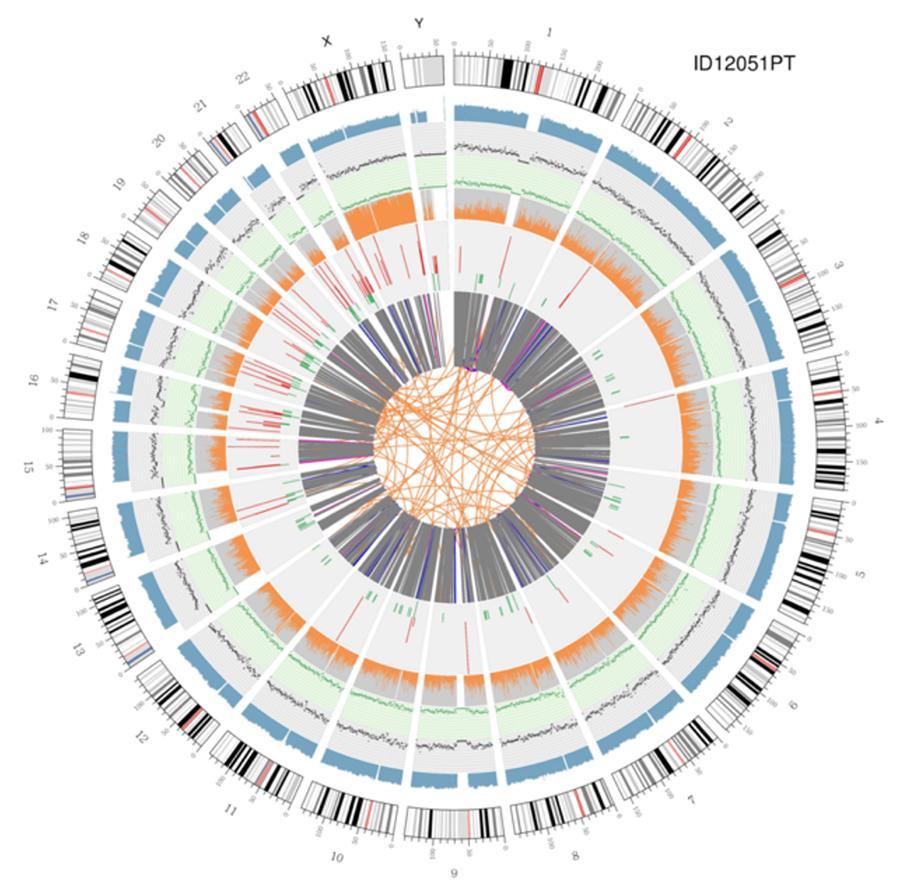
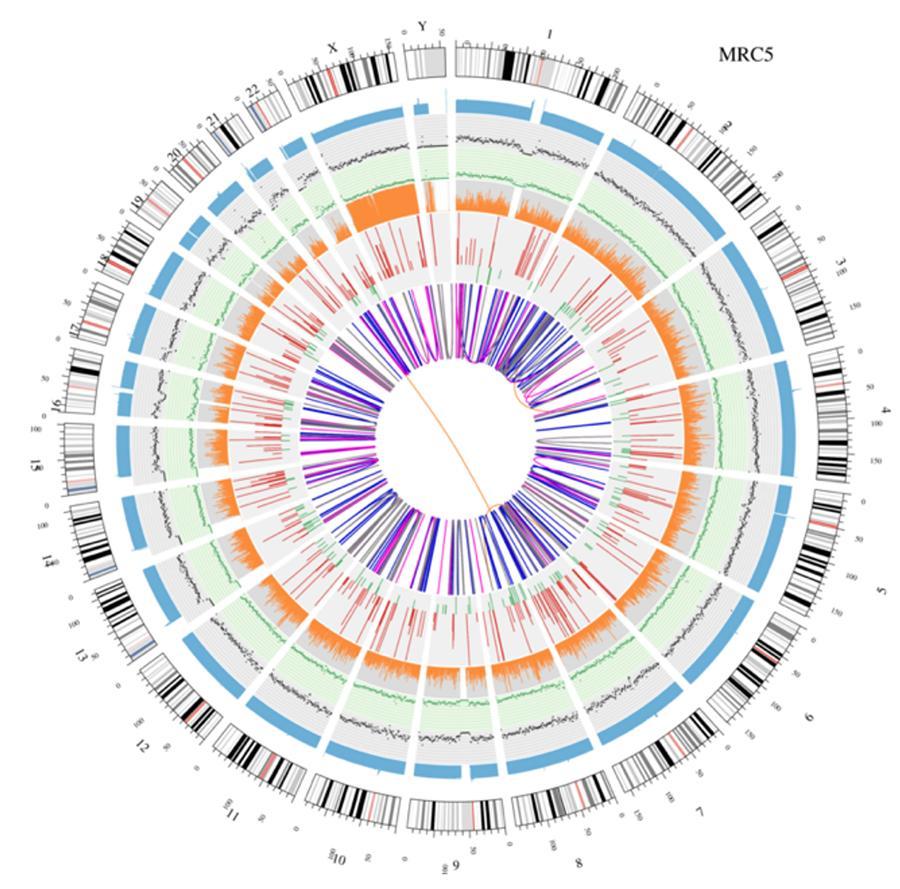
Bedeutung der verschiedenen konzentrischen Kreise
- Der äußere Kreis (der erste Kreis) ist die Chromosomenzahl.
- Der zweite Ring (blau) repräsentiert die Abdeckung von Lesevorgängen im Histogrammstil. Jedes Histogramm ist die durchschnittliche Abdeckung einer Fläche von 0,5 Mbit / s.
- Der dritte Ring (schwarz) repräsentiert die Dichte der INDELs in einem "Dispersionsgraphen" -Stil. Jeder Schwarzpunkt wird als Anzahl der INDELs (kleine Einfügungen / Löschungen) in einem Bereich von 1 Mbit / s berechnet.
- Der vierte Ring (grün) repräsentiert die Dichte snp in einem "Dispersionsgraphen" -Stil. Jeder grüne Punkt wird als Anzahl von SNPs (Einzelnukleotidvarianten) in einem Bereich von 1 Mbit / s berechnet.
- Der fünfte Ring repräsentiert den Anteil von Homozygotie (orange) und Heterozygotie (grau) SNP im Histogrammstil. Jedes Histogramm wird aus einer 1-Mbit / s-Region berechnet.
- Der sechste Ring repräsentiert die Inferenz von CNV (Varianten in Anzahl der Kopien). Rot bedeutet, DNA-Stücke zu gewinnen, und Grün bedeutet Verlust.
- Der zentralste Ring repräsentiert die Inferenz von SV (Strukturvarianten) in den exonischen und Spleißbereichen. ZWISCHEN (orange, Translokationen), INS (grün, Insertionen), DEL (Deletionen, grau), DUP (Duplikationen, pink) und INV (Inversionen, blau).
Schlussfolgerungen
Die beiden Genome zeigen wichtige Unterschiede. Insbesondere das Impfstoffgenom ist im Vergleich zu dem der 1966 hinterlegten Zelllinie stark verändert. Es muss gesagt werden, dass wir nicht die Möglichkeit hatten, die Zelllinie zu sequenzieren, die von GlaxoSmithKline für die Herstellung des Priorix Tetra-Impfstoffs verwendet wird, sondern die des Bestandes Original. Pharmaunternehmen, die Zelllinien zur Herstellung von Arzneimitteln und Impfstoffen verwenden, haben ihre MRC-5-Linie, aber dies ändert nichts an unserem Verdacht, dh der Tatsache, dass keine genetische Stabilitätskontrolle in regelmäßigen Abständen erforderlich ist, und dies ist unserer Meinung nach sehr ernst , vor allem, weil es unbegrenzt präsent sein darf.

