Vaccinegate: MRC-5 in Priorix Tetra - Volledige genoomsequencing

Deze nieuwste analyses werden mogelijk gemaakt dankzij de actieve bijdrage van de Franse verenigingen Vereniging Liberté Informatie Santé (ALIS), Ligue nationale pour la liberté des vaccinaties (LNPLV) en de Australische vereniging Australian Vaccination-risk Network (AVN), dat bedanken we.
Sequencing van de nieuwe generatie is het geprefereerde instrument geworden voor diepgaande analyse op het gebied van biologie en medische wetenschap, met name hoge precisie. Dankzij deze tools kunnen we een aantal toepassingen op een modernere en meer uitgebreide manier benaderen, zoals de novo sequencing, metagenomische en epigenomische studies, transcriptoomsequencing en genoom-re-sequencing.
Deze laatste (re-sequencing) wordt grotendeels op menselijk gebied gebruikt, zowel voor onderzoeks- als diagnostische doeleinden en bestaat uit NGS - Next Generation Sequencing van een heel enkel genoom, om de Single Nucleotide-mutaties (SNP), inserties en deleties van meer of minder lange sequenties die zijn opgetreden op bepaalde locaties van het genoom, en variaties in het aantal kopieën van genomische delen / genen (CNV, Copy Number Variants).
Deze procedure helpt om het ontwikkelingsmechanisme van sommige pathologieën te begrijpen, om de aanwijzingen voor een toekomstige klinische behandeling te identificeren, zoals bijvoorbeeld in het geval van kanker. Door deze methode kan het genetische erfgoed van een kankerpatiënt inderdaad volledig worden gedecodeerd in zowel normaal als kankerweefsel, waardoor we kunnen begrijpen wat er precies is veranderd binnen het genoom en, indien mogelijk, hoe kan worden ingegrepen met gerichte maatregelen.
De sequentiebepalingsprocedure vereist dat het DNA van een individu mechanisch wordt gebroken in fragmenten van kleine afmetingen (400-500 basenparen) en dat kunstmatige DNA-delen, adapters genaamd, aan deze fragmenten worden gebonden; adapters maken het mogelijk om de menselijke DNA-fragmenten te binden aan een glazen oppervlak waarop de basismeting (A, C, G, T) wordt uitgevoerd. Het aflezen van de DNA-basenparen vindt plaats door middel van chemische reacties, namelijk het inbouwen van nucleotiden die zijn gemarkeerd door fluorescerende moleculen. De miljoen aldus verkregen sequenties (reads) worden vervolgens in kaart gebracht op het menselijke referentiegenoom door specifieke software en alle varianten worden geïdentificeerd door het geanalyseerde genoom te vergelijken met het referentiegenoom.
Dezelfde procedure is uitgevoerd op het menselijk genoom in Priorix® Tetra lot n. A71CB256A, genoom dat behoort tot cellijn MRC-5 (van foetale oorsprong); het werk is uitgevoerd door een bedrijf in de VS, dat zich routinematig bezighoudt met analyse van de sequentiebepaling van het menselijk genoom. *
* de naam van het laboratorium dat de analyse heeft uitgevoerd, wordt opgenomen in de volgende formele klacht die we zullen indienen bij de openbare aanklager van Rome en ook bij de Italiaanse en Europese regelgevende instanties. De verenigingen die de door Corvelva gefinancierde analyse indienen, zullen ook onmiddellijk op de hoogte worden gehouden van deze schokkende resultaten. We kunnen niet ontkennen dat we, vooral als ouders, bedroefd zijn over de resultaten die we rapporteren - alsof wat we tot nu toe hebben ontdekt niet genoeg was om ons zorgen over te maken.
Resultaten
Het humane referentiegenoom bleek overeen te komen met 99.76% afgelezen uit vaccin-DNA, wat bijna in zijn geheel betekent. Het menselijke foetale DNA dat in dit vaccin wordt gepresenteerd, is een enkel heel genoom, wat betekent dat het vaccin genomisch DNA bevat met alle chromosomen van een mannelijk individu (MRC-5 is in feite afkomstig van een mannelijke foetus).
Hieronder worden de analyseresultaten van verschillende soorten varianten vergeleken met het menselijke referentiegenoom.
Variant met één nucleotide (SNP - polymorfisme met één nucleotide) en korte inserties / deleties (InDels)
Varianten van DNA-enkele basen (SNP) zijn polymorfismen, wat genetische materiaalmutaties van een enkel nucleotide betekent.
De 'InDels' zijn in plaats daarvan kleine invoegingen en deleties van minder dan 50 bp en vormen een andere klasse van genomische varianten in het menselijke genoom.
In het vaccingenoomgenoom zijn 3.6 miljoen SNP geïdentificeerd (waarvan 98.31% al is gerapporteerd in de openbare database dbSNP en 61.805 nieuw, dat betekent origineel in dit DNA) en ongeveer 804.000 InDels (waarvan 89.42% al is gerapporteerd in dbSNP en 85.106 nieuw).
De hoeveelheid SNP komt overeen met wat in de literatuur is gerapporteerd over / in "typisch menselijk genoom", terwijl de InDels resulteert in een grotere hoeveelheid vergeleken met wat is gerapporteerd door "The 1000 Genomes Project Consortium", namelijk 800.000 vergeleken met 600.000 .
CNV (kopie nummer varianten) en SV (structurele varianten)
De kopieaantalvarianten (CNV's) zijn genomische varianten vanwege variaties in het aantal kopieën van relatief grote fragmenten (langer dan 50 bp) tussen individuele genomen. Er zijn twee soorten CNV's: type "gain" (winst van kopieën) en type "loss" (verlies van kopieën). 218 CNV's werden gedetecteerd in het humane vaccingenoom, waarvan 82 "winst" (die een deel van het genoom van ongeveer 6.9 miljoen basenparen bedekken) en 136 CNV's van het "verlies" type (die een deel van het genoom van ongeveer 70 bedekken) miljoen bases).
Zoals beschreven door The 1000 Genomes Project Consortium in "Een wereldwijde referentie voor menselijke genetische variatie (Nature, vol. 526, 10 oktober 2015)" bevat een typisch menselijk genoom 2,100 tot 2,500 grote varianten, waaronder:
- 1,000 grote verwijderingen
- 160 varianten in aantal exemplaren (CNV's)
- 10 inversies
Die samen de 20 miljoen basen van de reeks beïnvloeden, ook rekening houdend met de invoegingen.
Zoals gezien voor de korte INDEL's, zelfs in het geval van grote inserties en deleties, is het vaccingenoom daarom niet in overeenstemming met een "normaal" menselijk genoom, omdat het veel meer "herschikt" is dan een genoom van een gewone persoon.
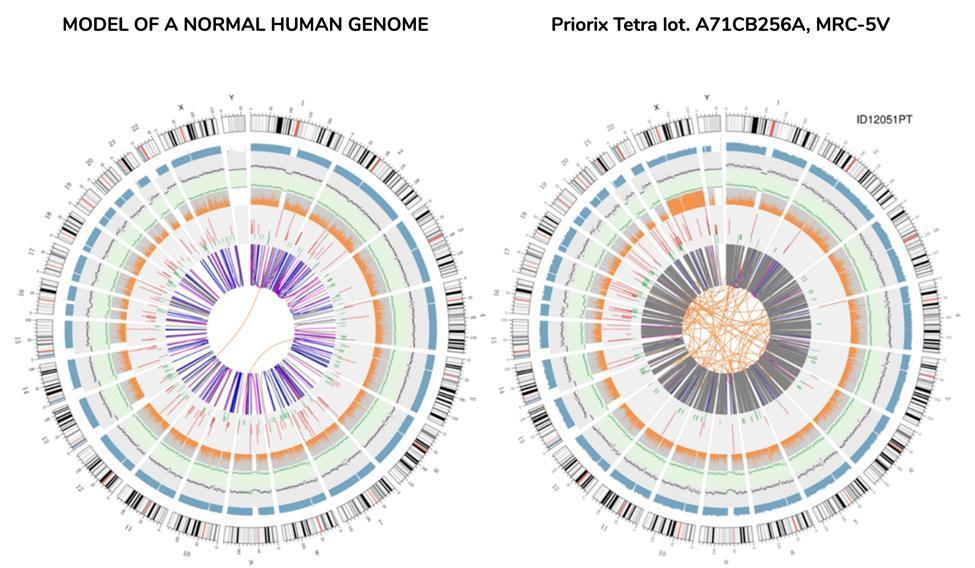
Circulaire visie van genoom (circosplot)
Een grafische weergave van het vaccingenoom genaamd "circosplot" (gewoonlijk gebruikt om een opnieuw bepaald genoom weer te geven), wordt hieronder weergegeven, naast een andere die een genoom weergeeft waarvan de sequentie is bepaald uit DNA dat is geëxtraheerd uit bloed van een gezond individu - "normaal" genoom :
Betekenis van de concentrische cirkels
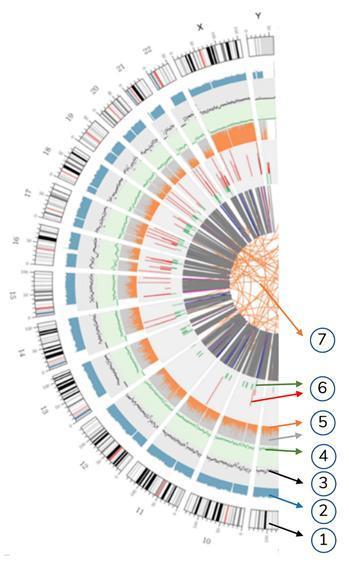
7) De meest centrale ring vertegenwoordigt de SV (structurele varianten) inferentie in de exon- en splitsingsgebieden. TRA (oranje, translocaties), INS (groen, invoegingen), DEL (verwijderingen, grijs), DUP (duplicaties, roze) en INV (inversies, blauw).
6) de zesde cirkel vertegenwoordigt de CNV-inferentie (varianten met aantal kopieën). Rood betekent DNA-sequenties winst en groen betekent verlies.
5) De vijfde ring vertegenwoordigt de SNP-verhouding in homozygositeit (oranje) en heterozygositeit (grijs) in histogramlay-out.
4) De vierde ring (groen) vertegenwoordigt de SNP-dichtheid in de indeling van de spreidingskaart.
3) De derde ring (zwart) vertegenwoordigt de dichtheid van de INDEL in de indeling van de spreidingskaart.
2) De tweede ring (lichtblauw) vertegenwoordigt de leesafdekking in de histogramlay-out.
1) De externe cirkel (de eerste cirkel) is het chromosoomnummer.
Een geschatte vergelijking kan ook worden gemaakt tussen foetaal DNA en het HeLa-cellen-DNA, de onsterfelijk gemaakte cellijn die ook wordt gebruikt om het poliovaccin te produceren.
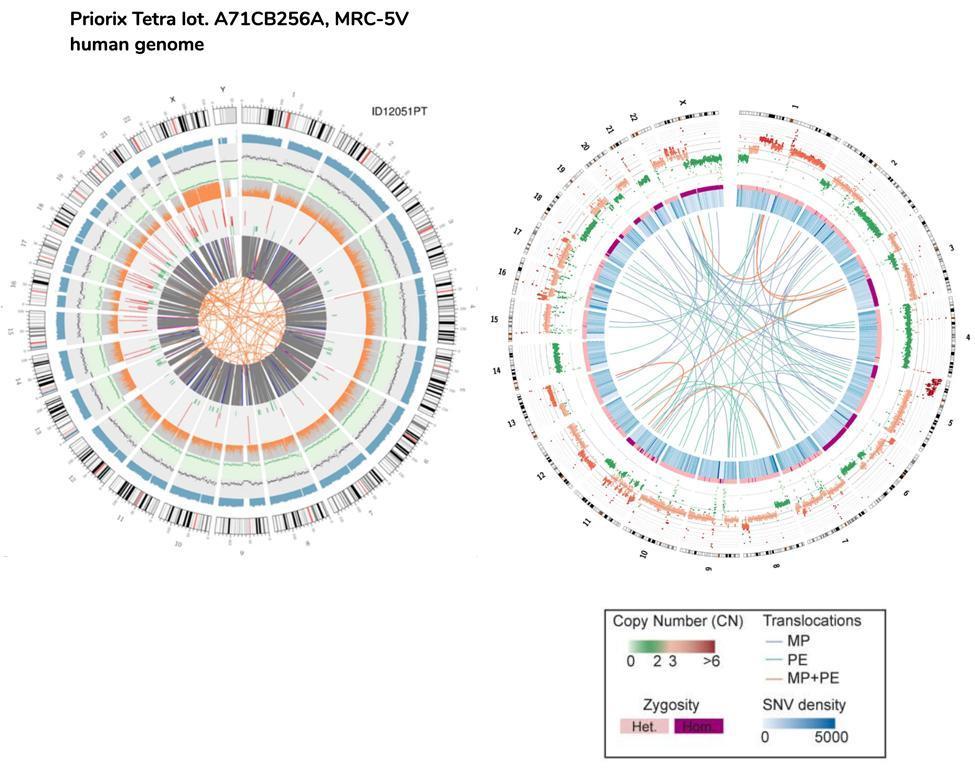
Merk op dat de HeLa-cellen translocaties die in de circosplot worden weergegeven door de lijnen van de kern, worden verwezen naar het hele genoom (vandaar het coderende en niet-coderende deel), terwijl ze in het geval van foetale vaccinatiecellen alleen verwijzen naar coderende genen .
Het is niet nodig om een wetenschapper te zijn om vanuit de circulatie in één oogopslag te begrijpen dat het vaccingenoom geen genoom is dat als "normaal" kan worden gedefinieerd. De oranje lijnen die in het midden van de circussen zijn verweven, niet zo talrijk in de corresponderende ring van het "normale" genoom, zijn al logisch voor de anomalie van dit genoom.
Variantanalyse in kankergenen
De analyse van SNP-, InDels-, CNV- en SV-varianten op 560 genen die zijn gekozen omdat ze betrokken zijn bij verschillende vormen van humane kanker, toont de aanwezigheid van talloze 'originele' varianten aan, dat wil zeggen dat ze niet eens aanwezig zijn in openbare databases, dus niet bekend in de literatuur. Met andere woorden, voor alle 560 geverifieerde genen zijn belangrijke modificaties van genen waarvan bekend is dat ze geassocieerd zijn met verschillende tumorvormen; bovendien zijn er varianten waarvan de gevolgen niet bekend zijn, maar die echter van invloed zijn op genen die betrokken zijn bij de inductie van menselijke kanker.
Conclusie
Het menselijke genomische DNA in het Priorix-lotvaccin. n. A71CB256A is duidelijk abnormaal en vertoont belangrijke inconsistenties in vergelijking met een typisch menselijk genoom, dat wil zeggen dat van een gezond mens. Er zijn verschillende onbekende varianten (niet vermeld in openbare databases) en sommige daarvan bevinden zich in genen die bij kanker betrokken zijn. Wat ook blijkbaar afwijkend is, is de overmaat aan genoom die veranderingen in het aantal kopieën (CNV) en structurele varianten (SV) vertoont, zoals translocaties, inserties, deleties, duplicaties en inversies, waarvan er vaak genen bij betrokken zijn.
De mogelijke bijdrage van de vele varianten (niet aanwezig in de wetenschappelijke literatuur en in openbare databases) aan het fenotype van de cellen die worden gebruikt voor de groei van vaccinvirussen is niet bekend.
In de PDF vindt u alle inzichten die ook betrekking hebben op de implicaties voor de begroeting en de correspondentie met de EMA.
Download link: CORVELVA-MRC-5-contained in Priorix-Tetra-Complete-genoom-sequencing

