Vaccinegate: complete genoomsequencing van MRC-5 in Priorix Tetra

Deze laatste analyses waren mogelijk dankzij de actieve bijdrage van de Franse verenigingen Vereniging Liberté Informatie Santé (ALIS), Ligue nationale pour la liberté des vaccinaties (LNPLV) en de Australische vereniging Australian Vaccination-risk Network (AVN) dat we bedanken.
De nieuwe generatie sequencers zijn uitgelezen instrumenten voor diepgaande analyses op het gebied van biologie en geneeskunde, met name de precisie. Deze tools maken een nieuwe en meer globale benadering mogelijk van een reeks toepassingen zoals de novo sequencing, metagenomics studies, epigenomics, transcriptome sequencing en genome re-sequencing.
Deze laatste toepassing (re-sequencing) is wijdverbreid in het menselijk veld, zowel voor onderzoeks- als diagnostische doeleinden en bestaat uit de sequencing met NGS (Next Generation Sequencing) -technologie van een volledig individueel genoom om single nucleotide mutations (SNP, uitspraak 'snip'), invoegingen en deleties van min of meer lange sequenties traden op in bepaalde posities van het genoom en variaties in het aantal kopieën van delen van genoom / genen (CNV, Copy Number Variants).
Deze procedure is nuttig om de mechanismen van de ontwikkeling van sommige pathologieën te begrijpen om de aanwijzingen voor toekomstige klinische behandeling te identificeren, zoals in het geval van kanker. Met deze methode kan het genetische erfgoed van een kankerpatiënt in feite volledig worden gedecodeerd in normaal en tumorweefsel, waardoor het mogelijk wordt te begrijpen wat er is veranderd in het genoom en, indien mogelijk, in te grijpen met gerichte protocollen.
De re-sequencing procedure vereist dat het DNA van een individu mechanisch wordt gebroken in kleine fragmenten (400-500 basenparen) en dat fragmenten worden gebonden aan kunstmatige DNA-kanalen die adapters worden genoemd, waarmee de fragmenten van Menselijk DNA op een glazen oppervlak waarop de basen worden afgelezen (A, C, G, T). De DNA-basen worden afgelezen door chemische reacties van nucleotiden gemerkt met fluorescente moleculen op te nemen. De miljoenen sequenties (leest) verkregen door sequentiëring op het glasoppervlak, worden vervolgens in kaart gebracht op het menselijke referentiegenoom met geschikte software en daarom worden alle varianten aanwezig in het geanalyseerde genoom geïdentificeerd, met betrekking tot de referentie.
Dezelfde procedure werd uitgevoerd op het menselijke genoom dat aanwezig is in Priorix Tetra-lot. n. A71CB256A, genoom behorend tot de MRC-5 cellijn (van foetale oorsprong); het werk werd uitgevoerd bij een bedrijf in de VS, dat routinematig de analyse van menselijke genomen behandelt door re-sequencing. *
* De naam van het laboratorium dat deze analyse heeft uitgevoerd, zal worden opgenomen in de volgende verklaring die we zullen deponeren bij het Openbaar Ministerie in Rome en bij de Italiaanse en Europese controle-instanties. De realiteiten die de resultaten neerleggen van de door Corvelva gefinancierde analyses zullen ook onmiddellijk worden bijgewerkt met deze raadselachtige resultaten. We ontkennen niet dat we als ouders in de eerste plaats verontrust zijn door de resultaten die we hieronder rapporteren - als wat we tot nu toe hebben ontdekt niet genoeg was.
resultaten
Het humane referentiegenoom bleek voor 99.76% te zijn bedekt met lezingen afkomstig van het vaccin-DNA, dus voor bijna het geheel. Het foetale menselijke DNA dat in dit vaccin wordt weergegeven, is daarom een compleet individueel genoom of is aanwezig in het genomische DNA-vaccin van alle chromosomen van een mannelijk individu (en in feite is de foetus waaruit de MRC-5-cellijn afkomstig is mannelijk).
Hieronder staan de resultaten van de analyse van de verschillende soorten varianten met betrekking tot het menselijke referentiegenoom.
Varianten met één nucleotide (SNP) en korte inserties / deleties (InDels)
De varianten van individuele DNA-basen (SNP, uitgesproken als 'snip') zijn polymorfismen, dat wil zeggen variaties van het genetische materiaal, gedragen door een enkel nucleotide. De 'InDels' zijn daarentegen kleine inserties en deleties van minder dan 50 pb lang en vormen een andere klasse van genomische varianten in het menselijk genoom.
Er zijn in totaal ongeveer 3.6 miljoen SNP's geïdentificeerd in het humane vaccingenoom (waarvan 98.31% al in de openbare database dbSNP en 61.805 nieuw of origineel van dit DNA werd gerapporteerd) en ongeveer 804 duizend InDels (waarvan 89.42% al gerapporteerd in dbSNP en 85.106 nieuw).
De hoeveelheid SNP is in overeenstemming met wat in de literatuur wordt gerapporteerd in een "typisch menselijk genoom", terwijl de indels in een grotere hoeveelheid zijn dan wat wordt gerapporteerd door het "The 1000 Genomes Project Consortium" of 800 duizend vergeleken met 600 duizend.
CNV (kopie nummer varianten) en SV (structurele varianten)
Varianten in aantal exemplaren (CNV) zijn genomische varianten vanwege variaties in het aantal exemplaren van relatief grote fragmenten (langer dan 50 bp) tussen individuele genomen. Er zijn twee soorten CNV: type "gain" (winst van kopieën) en type "loss" (verlies van kopieën). 218 CNV's zijn gedetecteerd in het humane vaccingenoom, waarvan 82 van het "gain" -type (met een gedeelte van het genoom gelijk aan ongeveer 6.9 miljoen basenparen) en 136 "CNV's" van het "verlies" -type (die een deel van het genoom dekken) ongeveer 70 miljoen bases).
Zoals beschreven door The 1000 Genomes Project Consortium in "Een wereldwijde referentie voor menselijke genetische variatie (Nature, vol. 526, 10 oktober 2015)" bevat een typisch menselijk genoom 2.100 tot 2.500 grote varianten, waaronder:
- 1.000 grote verwijderingen
- 160 variaties in aantal kopieën (CNV's)
- 10 inversies
die in het algemeen, ook rekening houdend met de invoegingen, invloed hebben op 20 miljoen sequentiebases.
Zoals waargenomen voor korte INDEL's, zelfs in het geval van grote inserties en deleties, is het vaccingenoom daarom niet in overeenstemming met een 'normaal' menselijk genoom, omdat het veel meer "herschikt" is dan een genoom van een gewoon individu.
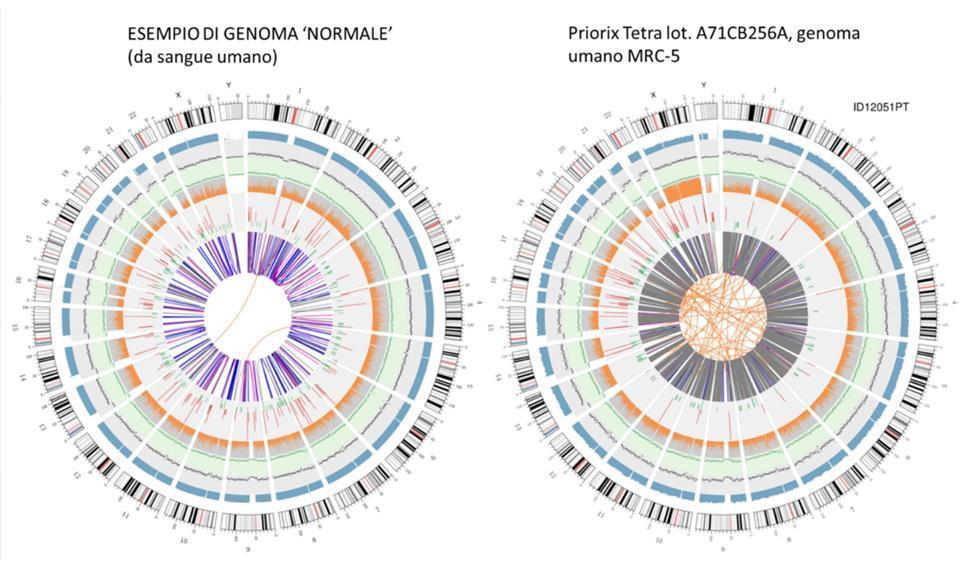
Circulaire visualisatie van het genoom (circosplot)
Een grafische weergave van het vaccingenoom, "circosplot" genoemd (dat gewoonlijk wordt gebruikt om een opnieuw bepaald genoom weer te geven), wordt hieronder weergegeven, naast een andere die een opnieuw bepaald genoom voorstelt, beginnend met DNA dat uit bloed is geëxtraheerd van een gezond individu - "normaal" genoom:
Betekenis van de verschillende concentrische cirkels
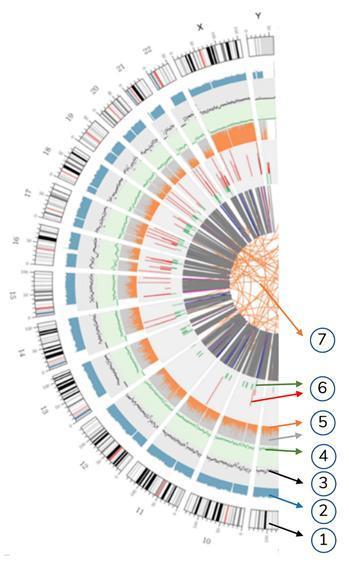
7) De meest centrale ring vertegenwoordigt de gevolgtrekking van SV (structurele varianten) in de exonische en splitsingsgebieden. TUSSEN (oranje, translocaties), INS (groen, invoegingen), DEL (verwijderingen, grijs), DUP (duplicaties, roze) en INV (inversies, blauw).
6) De zesde ring vertegenwoordigt de conclusie van CNV (varianten in aantal exemplaren). Rood betekent stukjes DNA verkrijgen en groen betekent verlies.
5) De vijfde ring vertegenwoordigt de verhouding van SNP in homozygositeit (oranje) en in heterozygositeit (grijs) in histogramstijl.
4) De vierde ring (groen) vertegenwoordigt de dichtheid snp in een "dispersiegrafiek" stijl.
3) De derde ring (zwart) vertegenwoordigt de dichtheid van de INDEL's in een "dispersiegrafiek" -stijl.
2) De tweede ring (blauw) geeft de dekking weer van histogram-achtige metingen.
1) De buitenste cirkel (de eerste cirkel) is het chromosoomnummer.
Een ruwe vergelijking kan ook worden gemaakt tussen foetaal DNA en HeLa-cel-DNA, de onsterfelijk gemaakte cellijn die ook wordt gebruikt bij de productie van het poliovaccin.
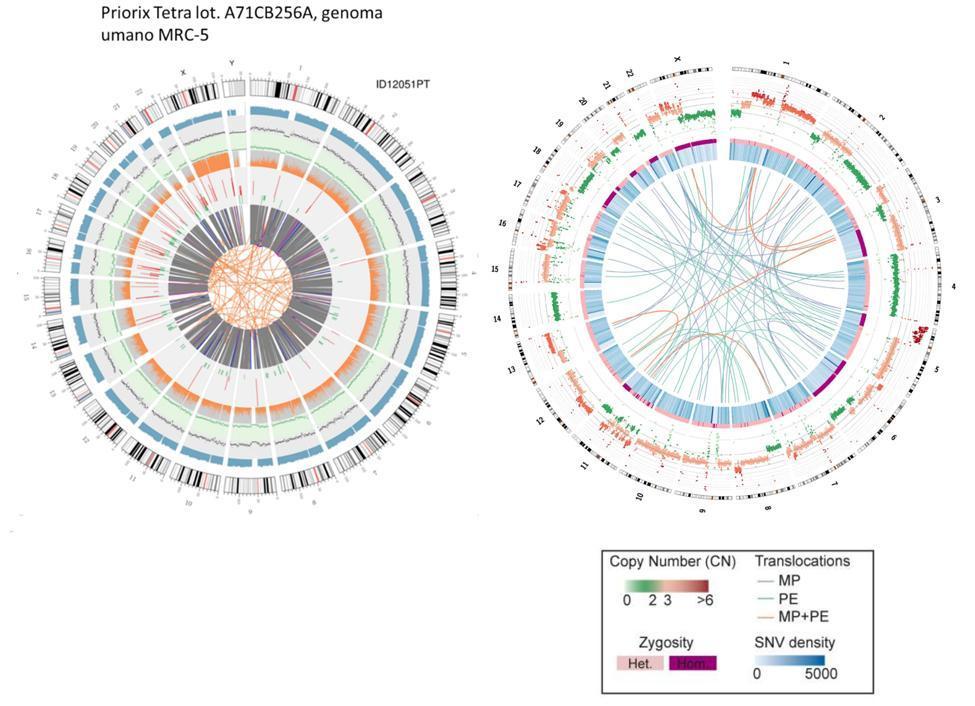
Er moet worden benadrukt dat de translocaties van de HeLa-cellen die in de circosplot worden weergegeven door de kernlijnen verwijzen naar het hele genoom (dus coderend en niet-coderend deel), terwijl ze in het geval van foetale vaccincellen alleen verwijzen naar de coderende genen.
U hoeft geen wetenschapper te zijn om vanuit de circus in één oogopslag te begrijpen dat het vaccingenoom geen genoom is dat als "normaal" kan worden gedefinieerd. De oranje lijnen die verweven zijn in het midden van de circo's, niet zo talrijk aanwezig in de corresponderende ring van het "normale" genoom, doen ons de anomalie van dit genoom al raden.
conclusies
Het menselijke genomische DNA in het Prorix-vaccin. n. A71CB256A is duidelijk abnormaal en vertoont belangrijke inconsistenties met betrekking tot een typisch menselijk genoom, dat wil zeggen dat van een gezond individu. Er zijn talloze onbekende varianten (niet genoteerd in openbare databases) en een aantal daarvan bevinden zich in genen die bij kanker betrokken zijn. Wat ook duidelijk abnormaal is, is het overtollige genoom dat veranderingen in het aantal kopieën (CNV) en structurele varianten (SV) vertoont, zoals translocaties, inserties, deleties, duplicaties en inversies, waarvan vele met genen.
De mogelijke bijdrage van de vele varianten (niet aanwezig in de wetenschappelijke literatuur en in openbare databases) aan het fenotype van de cellen die worden gebruikt voor de groei van vaccinvirussen is niet bekend.
In de PDF vindt u alle inzichten die ook betrekking hebben op de implicaties voor de gezondheid en correspondentie met de EMA.
Download link: CORVELVA-Ri-genoom-sequencing-humaan-Priorix-Tetra.pdf

